初めに
R0は基本再生産数といいます。Rtは実行再生産数といいます。
Rt = R0 × 封じ込めの効果 × 接触機会の削減効果
Rtは、重要な指標として重宝されています。WHOのreginal officeの資料に、その計算方法が説明されていました。要は、RのパッケージEpiEstimを使って計算してよということみたいです。
Pan American Health Organization. World Health Organization. Regional Office for the Americas:https://iris.paho.org/handle/10665.2/52405
データ
東京のコロナ感染者数データを使います。2020年1月24日~2021年6月23日まであります。
東京都サイト:https://catalog.data.metro.tokyo.lg.jp/dataset/t000010d0000000068/resource/c2d997db-1450-43fa-8037-ebb11ec28d4c
加工の概要は 日ごとに感染者数集計 → 欠損日の補完 です。以下に詳しく解説しています。
library(tidyverse)
df <- readxl::read_xlsx("130001_tokyo_covid19_patients.xlsx")
df <- as.data.frame(df$`公表_年月日`)
colnames(df) <- "date"
df <- df %>% group_by(date) %>% summarise(number=n())
date_df <- as.data.frame(data.frame(matrix(rep(NA, 1), nrow=1))[numeric(0), ])
colnames(date_df) <- c("date")
date_series <- seq(from=as.Date("2020/1/24", format="%Y/%m/%d"),
by =1,
to = as.Date("2021/6/23", format="%Y/%m/%d"))
for(i in 1:517){
date_df[i,1] <- as.character(date_series[i])
}
date_df$date <- as.Date(date_df$date,format="%Y-%m-%d")
df2 <- left_join(date_df,df,by="date")
for( i in 2:(nrow(df2)-1) ){
if ( is.na(df2$number[i]) ) {
if ( is.na(df2$number[i+1]) ){
df2$number[i] <- df2$number[i-1]
}else{
df2$number[i] <- (df2$number[i+1]+df2$number[i-1])/2
}
}
}
df2$date <- as.Date(df2$date, format="%Y-%m-%d")
df2 date number
1 2020-01-24 1.0
2 2020-01-25 1.0
3 2020-01-26 1.0それでは、さっそくEpiEstimを使ってRtを計算したいと思います。
計算の方法は、estimate_R(感染者数データ、method、、、mean_si、std_si) です。この関数に計算してもらうには、mean_si、std_siが必要です。siとは何でしょうか。
serial interval(si)とは、1人目が発症した日と、1人目が感染させた2人目が発症した日の間の間隔です。感染動態を知る上で非常に重要なパラメータです。
WHOの資料には、serial interval mean of 4.8 days and a standard deviation of 2.3.と書かれています。
method等の詳細は、パッケージ作者のサンプルを見てください。
https://cran.r-project.org/web/packages/EpiEstim/vignettes/demo.html
library(EpiEstim)
res_parametric_si <- estimate_R(df2$number,
method="parametric_si",
config = make_config(
list(mean_si = 4.8,
std_si = 2.3)
)
)
plot(res_parametric_si)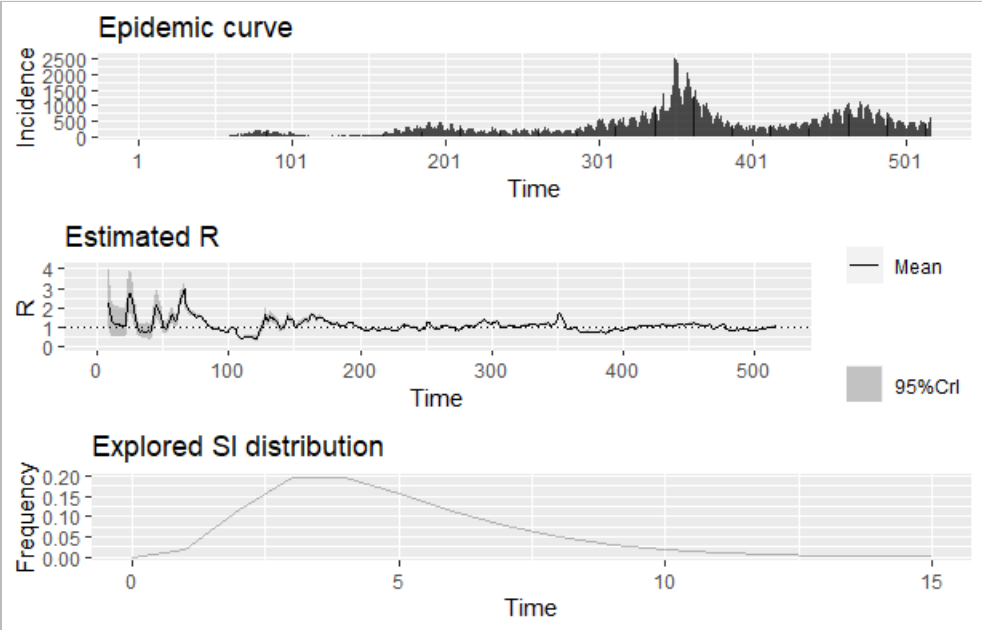
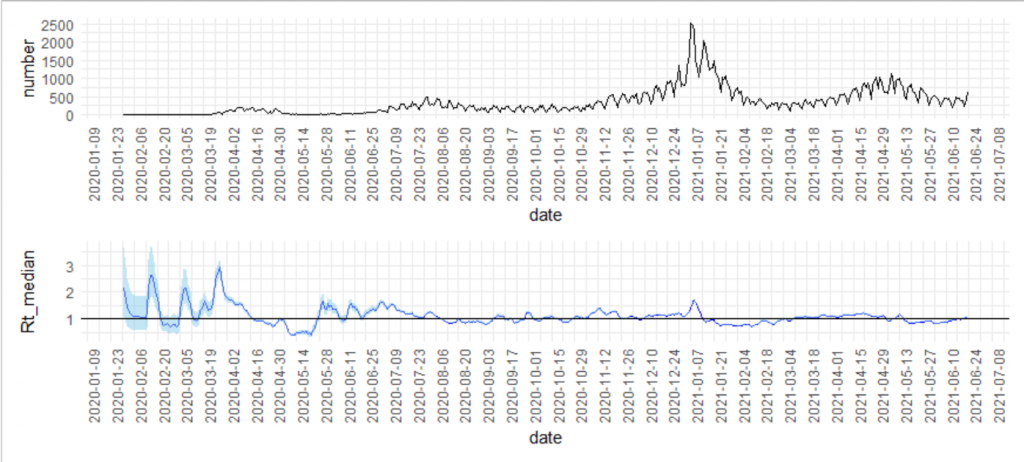
plotで一発で書けるのはいいのですが、軸がづれていて見ずらいです。ggplotとpatchworkで工夫します。
library(patchwork)
gg1 <- ggplot(df2 %>% tail(-7))+
geom_line(aes(x = date, y = number))+
theme_minimal()+
scale_x_date(date_breaks ="14 day")+
theme(axis.text.x = element_text(angle = 90))
gg2 <- tibble( df2 %>% tail(-7),
Rt_median = res_parametric_si$R$`Median(R)`,
Rt_quantile05 = res_parametric_si$R$`Quantile.0.05(R)`,
Rt_quantile95 = res_parametric_si$R$`Quantile.0.95(R)`
) %>%
ggplot()+
geom_line(aes(x = date, y = Rt_median),color="blue")+
geom_ribbon(aes(x=date, y = Rt_median,
ymax = Rt_quantile95,
ymin = Rt_quantile05),
alpha = 0.5, fill="skyblue")+
geom_hline(yintercept =1)+
theme_minimal()+
scale_x_date(date_breaks ="14 day")+
theme(axis.text.x = element_text(angle = 90))
gg1/gg2 
結構、簡単に計算できますね!とは言っても、serial interval meanとsd の情報がないと計算できないですね。もし、感染初期だったら具体例を観察して推測するんだと思います。そんなにデータ取るのは難しくはないと思われます。
