血液検査は、ある値を境に病気である・ないを分ける基準値が設定されています。その値をカットオフ値と言います。
ただ、獣医師は数値のみを見て機械的に病気、病気ではないと判断するわけではありません。ほかの検査や臨床症状を総合的に判断して、病気と診断します。なぜならば、検査が常に病気を正確に診断してくれるわけではないからです。
今回は、カットオフ値について知識を深めるために研究したいと思います。
使うライブラリはpROCです。その他、グラフの描写のためにtidyverseとRColorBrewerを読み込みます。
想定としては、病気のグループと、非病気のグループを2グループを想定します。グループは100個体からなり、実験により検査Aが実施され、検査数値のデータを取得したと仮定します。
病気グループは、平均10, SD = 7の正規分布からランダムに数値を100個取得しました。
非病気グループは、平均5, SD = 5の正規分布からランダムに数値を100個取得しました。まず、グラフ化してみます。
library(tidyverse)
library(pROC)
library(RColorBrewer)
set.seed(1469)
sick <- rnorm(100, mean = 10, sd = 7)
set.seed(1469)
normal <- rnorm(100, mean= 5, sd = 5)
sick <- tibble(sick)
names(sick) <- "score"
sick$label <- "sick"
normal <- tibble(normal)
names(normal) <- "score"
normal$label <- "normal"
sn <- rbind(sick, normal)
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()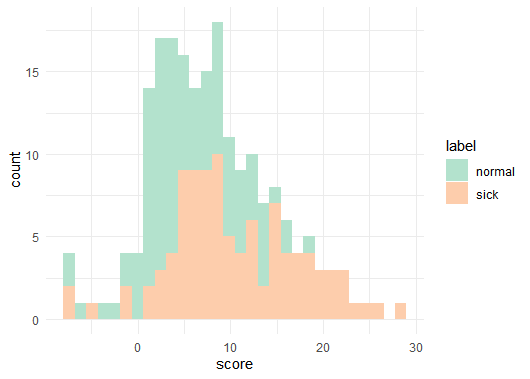
病気の個体と、病気ではない個体の数値の分布がかなり重なっています。平均に差はあるのでしょうか。
set.seed(1469)
sick <- rnorm(100, mean = 10, sd = 7)
set.seed(1469)
normal <- rnorm(100, mean= 5, sd = 5)
t.test(sick,normal)p-value =1.733e-08 なので有意水準5%では有意差があります。
統計的に差があると言っても、データをグラフで見ると病気と非病気グループには差がないように見えます。
ROCカーブを使って、カットオフ値を探します。まずはROCcurveを描きます。
sn$label <- factor(sn$label)
ro <- roc(response =sn$label,
predictor = sn$score)
plot(ro,
identity = TRUE,
print.thres = "best",
print.thres.best.method="closest.topleft"
)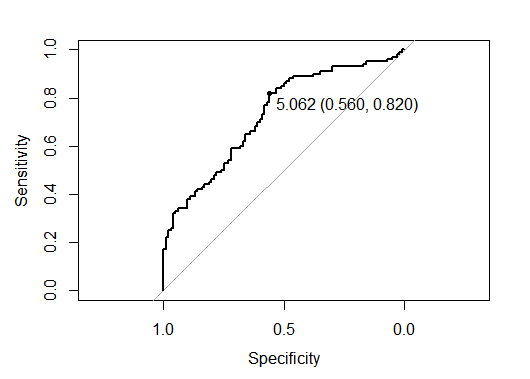
横軸にSpecificity、縦軸にsensitivityがあります。個体のデータに検査scoreがありますが、この数値のカットオフ値をずらしたときに、specificityとsensitivityがどうなるかをグラフ化したものがROCです。
グラフ上に5.062(0.560, 0.820)という数字が書かれています。この数値は、sensitivityとspecificityが両方高くなる左上に一番近い点のscoreのカットオフ値、specificity、sensitivityです。print.thres.best.method=”closest.topleft”というのが、その点の探索方法です。
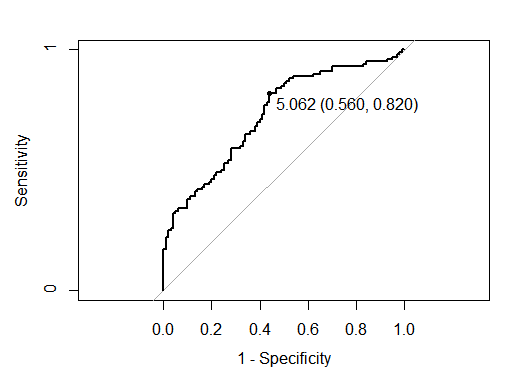
教科書には横軸が1-Specificityになっているかもしれません。その場合は、以下のようにlegacy.axes = TRUEを追加すると同じものが書けます。
plot(ro,
identity = TRUE,
print.thres = "best",
print.thres.best.method="closest.topleft",
legacy.axes = TRUE
)
数値を詳しく見てみます。
best <- coords(ro,
x = "best",
ret="all",
best.method = "closest.topleft")
best| threshold | specificity | sensitivity |
| 5.061974 | 0.56 | 0.82 |
Thresholdがカットオフ値です。このカットオフ値を使うと、感度82%、特異度56%の検査になります。
例えば、クッシング症候群の検査である低用量デキサメタゾン抑制試験は、以下のような感度と特異性を持っているようです。
研究1- 感度100 : 特異度73
研究2- 感度100 : 特異度44
研究3- 感度96 : 特異度70
研究4- 感度85 : 特異度73
ACTH試験は、以下のような感度と特異性を持っているようです。
研究1- 感度89 : 特異度82
研究2- 感度80 : 特異度86
研究3- 感度94 : 特異度91
カットオフ値を入れてグラフ化してみます。
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
geom_vline(xintercept = best$threshold)+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()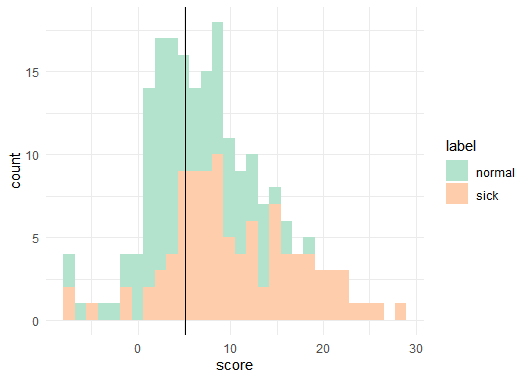
感度82%、特異度56%の検査ということだけ聞くと、私はふーん、まあいいかと思ってしまいそうです。データを視覚化すれば、あまりいい検査ではないことが分かります。視覚化することの重要性を感じます。
病気グループの数値の分布を少しだけ変化させてみます
平均10, SD = 7の正規分布 → 平均10, SD = 5の正規分布
set.seed(1513)
sick <- rnorm(100, mean = 10, sd = 5)
set.seed(1513)
normal <- rnorm(100, mean= 5, sd = 5)
sick <- tibble(sick)
names(sick) <- "score"
sick$label <- "sick"
normal <- tibble(normal)
names(normal) <- "score"
normal$label <- "normal"
sn <- rbind(sick, normal)
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()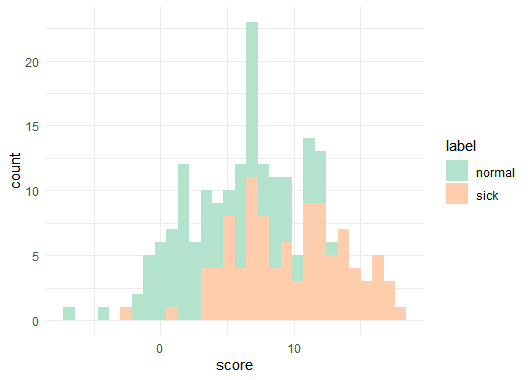
ROCカーブを描いてみます。今回は、カットオフ値を探索する方法をprint.thres.best.method=”youden”にします。左上の点に近いものを探す方法(closest.topleft)ではなく、slope1の直線から一番離れている点を探します。
sn$label <- factor(sn$label)
ro <- roc(response =sn$label,
predictor = sn$score)
plot(ro,
identity = TRUE,
print.thres = "best",
print.thres.best.method="youden")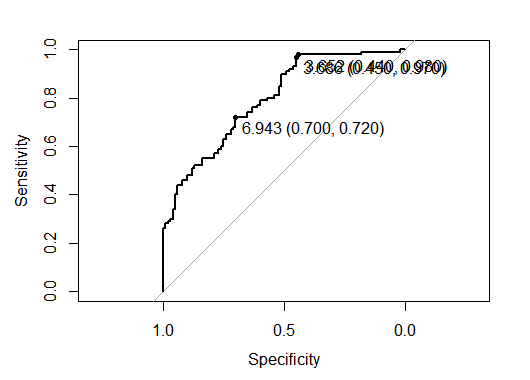
ベストポイントが3つあるようですね。ベストポイントの数値を見てみます。
best <- coords(ro,
x = "best",
ret="all",
best.method = "youden")
best| threshold | specificity | sensitivity |
| 3.651746 | 0.44 | 0.98 |
| 3.685732 | 0.45 | 0.97 |
| 6.943289 | 0.70 | 0.72 |
感度98%、特異度44%と聞くと、やはり私にはふーん、感度高いから偽陽性に注意しようと思うくらいです。修業がたりないでしょうか。視覚化してみます。
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
geom_vline(xintercept = best$threshold[1])+
geom_vline(xintercept = best$threshold[2])+
geom_vline(xintercept = best$threshold[3])+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()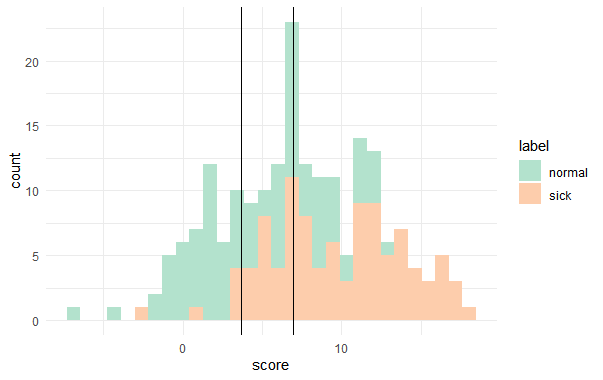
score 3.6のカットオフだと正常個体もほとんど陽性にしてしまいますね。これはまずい検査です。
参考:理想形
病気グループと非病気グループがはっきりと分かれている場合
set.seed(1469)
sick <- rnorm(100, mean = 15, sd = 1)
set.seed(1469)
normal <- rnorm(100, mean= 5, sd = 1)
t.test(sick,normal)
sick <- tibble(sick)
names(sick) <- "score"
sick$label <- "sick"
normal <- tibble(normal)
names(normal) <- "score"
normal$label <- "normal"
sn <- rbind(sick, normal)
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()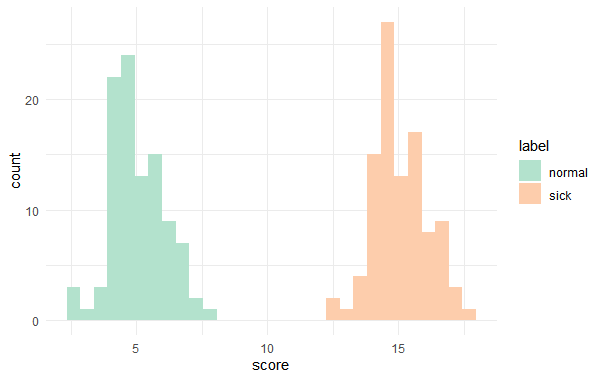
sn$label <- factor(sn$label)
ro <- roc(response =sn$label,
predictor = sn$score)
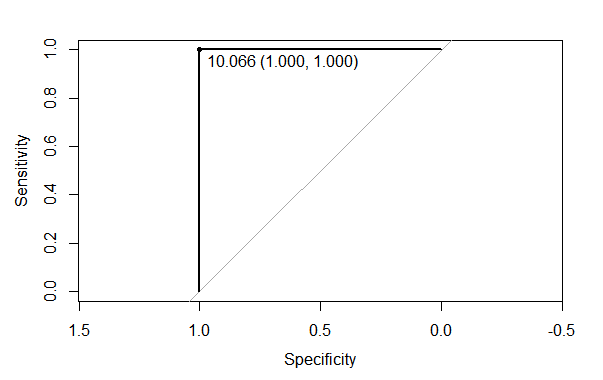
plot(ro,
identity = TRUE,
print.thres = "best",
print.thres.best.method="closest.topleft",
)
best <- coords(ro,
x = "best",
ret="all",
best.method = "closest.topleft")
ggplot(sn) +
geom_histogram(aes(x=score,fill=label))+
geom_vline(xintercept = best$threshold)+
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()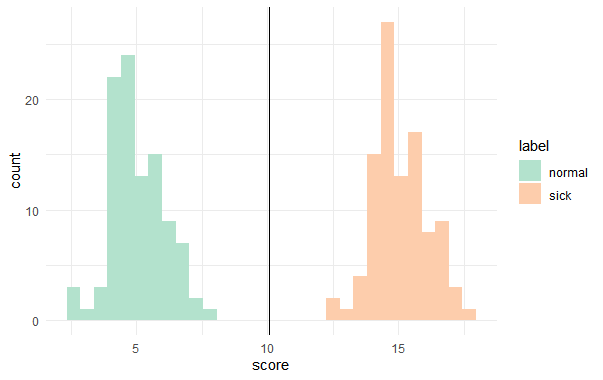
Decision Treeでカットオフ値を決める
この分野で、機械学習をつかってカットオフ値を決めるというのはあまり聞いたことがありません。私が知らないだけかもしれません。
決定木を使って、2つのグループを最も正確に分類できる分岐点を調べます。
library(rpart)
library(rpart.plot)
tree <- rpart(label ~ score, sn,
control = rpart.control(minsplit=1,
minbucket=1,
cp=0.1))
rpart.plot(tree)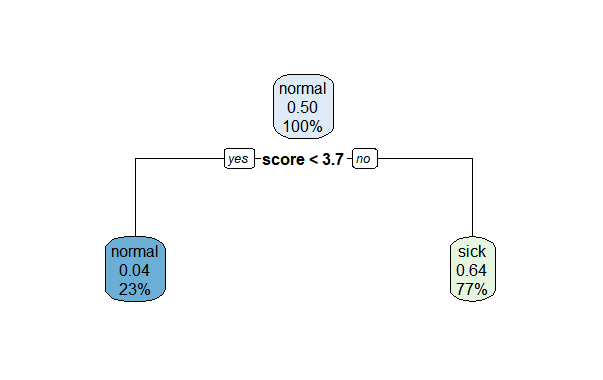
ROCと似たようなカットオフ値が分岐点になっています。細かい数値を見てみます。
print(tree)1) root 200 100 normal (0.50000000 0.50000000)
2) score< 3.651746 46 2 normal (0.95652174 0.04347826) *
3) score>=3.651746 154 56 sick (0.36363636 0.63636364) *
score 3.651746が分岐点だとわかります。これは、ROCのカットオフ値3.651746と全く同じですね!
計算方法は全く違うのに、同じ数字がでてきたことに驚きました。カットオフ値を探すためには、ROCで十分ってところですかね。
