教師なし学習は、kmeans と、hierarchical clusteringがメジャーなところです。今回は、hierarchical clusteringをRで実行してみます。
ライブラリ
ggdendro、dendextend、factoextraとtidyverseを使用します。
コード
Rのデフォルトデータirisを使用します。データは、変数の単位が揃ってようとスケーリングします。種類のデータは知らないことにします。そのためspeciesの列を除きます。
myiris <- iris %>% select(-Species)
myiris <- scale(myiris)
myiris <- as_tibble(myiris)hclustで、hierarchical clusteringを実行できます。その前にdistance matrixをつくらないといけません。あとで、distance matrixを詳しく見ますが、ここでは前処理だと思って下さい。
d <- dist(myiris)
clusters <- hclust(d,
method = "complete")methodの話はおいておいて、プロットしてみます。
plot(clusters)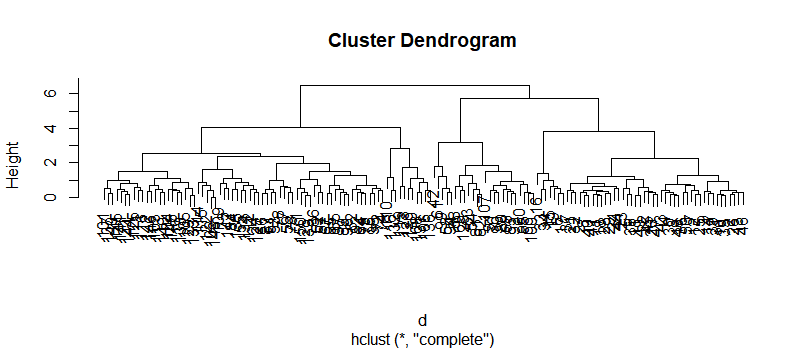
ggdendroで、少し見栄えを良くします。
ggdendro::ggdendrogram(clusters)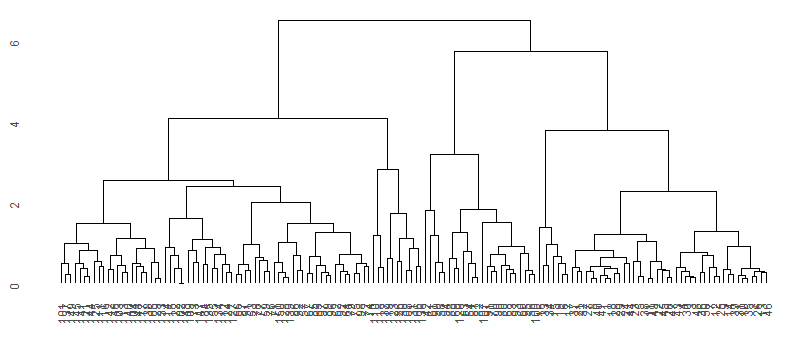
少しだけ見栄えが良くなりました。
ところで、hclustのmethodには、以下の項目が選択できます。linkageの方法です。
“ward.D”, “ward.D2”, “single”, “complete”, “average”, “mcquitty”, “median”, “centroid”
これらは、クラスター同士の距離計算の方法です。以下で、distance matrixをみてみましょう。
d2 <- dist(head(myiris))
d2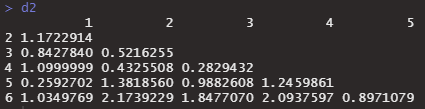
一番距離が短いのが0.259の1番と5番なので最初にくっつきます。では、くっついた後1-5番の点をどう扱うのでしょうか。そこがlinkage methodです。averageなら2つの平均だし、singleなら一番近い方、completeなら遠い方を採用して計算します。
2つのクラスター方法を比較する方法です。使い道はわかりませんが、かなり強そうな図が書けます。
clusters_complete <- hclust(d,
method = "complete")
clusters_average <- hclust(d,
method = "average")
library(dendextend)
dendlist(as.dendrogram(clusters_complete),
as.dendrogram(clusters_average)) %>%
tanglegram()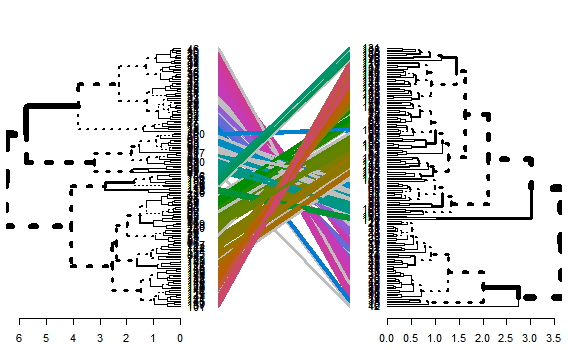
どこで伐採すべきか(グループ数の決め方)
いったい、いくつのグループに分けたらいいのでしょうか。3つのスコアで、最適なグループ数を見つけます。
肘法
factoextraパッケージのfviz_nbclustを使います。FUNclusterをhcutにします。ただ、hcutのデフォルトが以下の設定です。average法とか、complete法は設定できないようです。
Cluster method : ward.D2
Distance : euclidean
factoextra::fviz_nbclust(myiris,
FUNcluster = hcut,
method = "wss")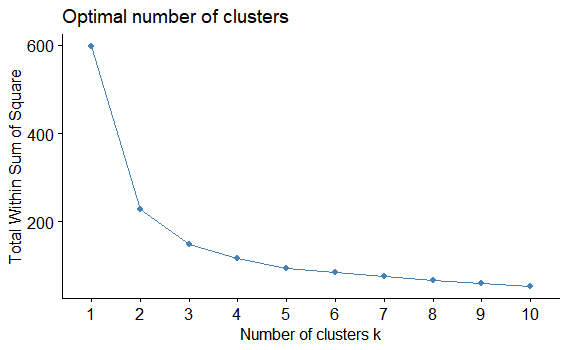
肘を曲げるところが最適な点なので、クラスター数は2です。
影絵法
factoextra::fviz_nbclust(myiris,
FUNcluster = hcut,
method = "silhouette")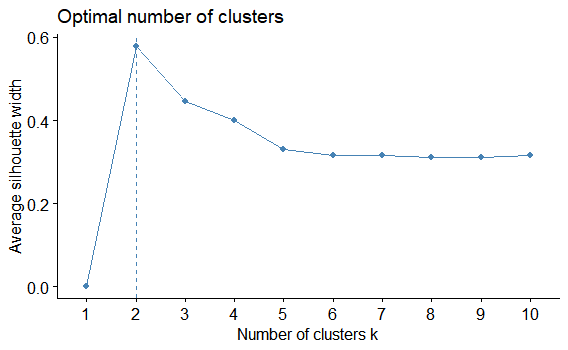
これは、シルエットの美しさポイントが一番高いところが選択ポイントになります。k=2です!
間法
factoextra::fviz_nbclust(myiris,
FUNcluster = hcut,
method = "gap_stat")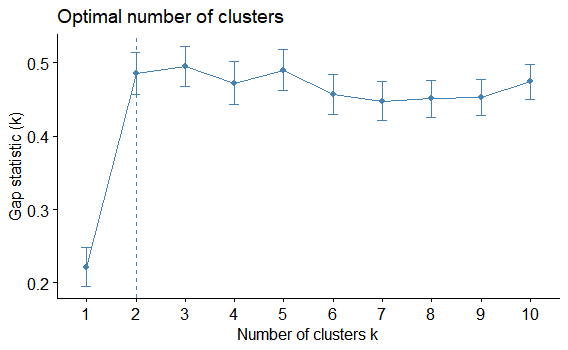
クラスターの最適数は2のようです。初めは、あれ?k=3じゃないのと思いました。この方法は、現在のKのgap statisticが、次のKのgap statisticの1標準偏差内となる最小のKを選択するようです。
クラスター数2で伐採することにします
cutreeで、伐採します。kでクラスター数を指定します。
d <- dist(myiris)
clusters_complete <- hclust(d,
method = "complete")
group <- cutree(clusters_complete, k=2)
head(group)> head(group) 111111
groupには、予想されたグループ名の列が入っています。
irisが4次元なので、仕方ないですが2変数だけを使ってプロットしてみます。
tibble(myiris, group = group) %>%
ggplot()+
geom_point(aes(x=Sepal.Length,
y=Sepal.Width,
color=as.character(group)))+
theme_minimal()+
labs(color = "Predicted Group")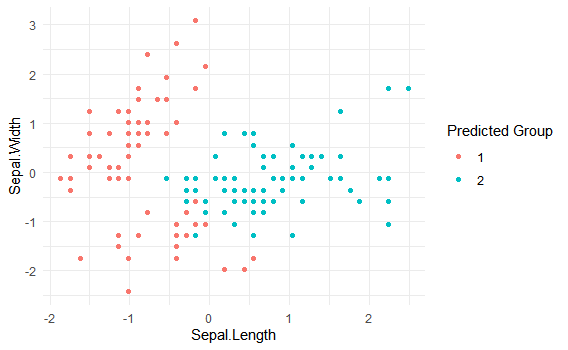
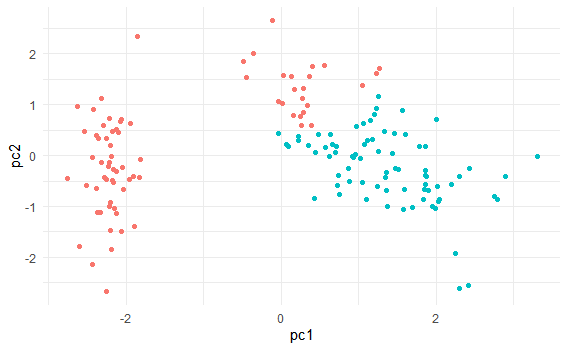
PCAを使って次元削減した図で表現すると以下のようになります。
pca <- prcomp(myiris, scale = F)
tibble(pc1 = pca$x[,1],
pc2 = pca$x[,2],
group = group) %>%
ggplot()+
geom_point(aes(x=pc1,
y=pc2,
color=as.character(group)))+
theme_minimal()+
theme(legend.position = "none")