教師なし学習は、kmeans と、hierarchical clusteringがメジャーなところです。今回は、kmeansをRで実行してみます。
ライブラリ
factoextraとtidyverseを使用します。
コード
まず、データはスケーリングした方がいいようです。列ごとに単位が同じでも平均が違う場合は数字のパワーバランスがおかしいので、問答無用でスケーリングしたいと思います。
使用するデータは、デフォルトのirisデータです。種類のデータは知らないことにします。そのためspeciesの列を除きます。
library(tidyverse)
myiris <- iris %>% select(-Species)
myiris <- scale(myiris)
myiris <- as_tibble(myiris)さっそくkmeansを実行します。kの数(centers)は、とりあえず2でやってみます。nstartは、最初のランダム状態を何回施行するかです。
kmeans2 <-kmeans(myiris,
centers = 2,
nstart = 50)
kmeans2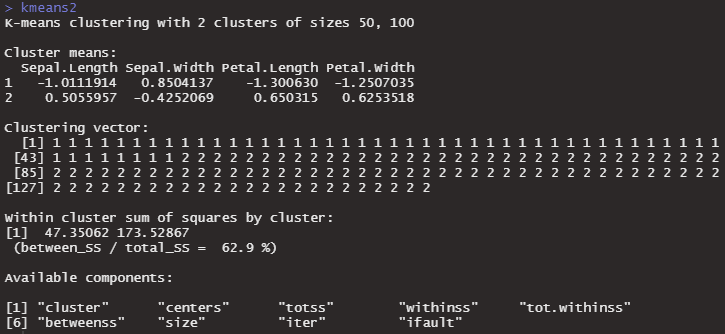
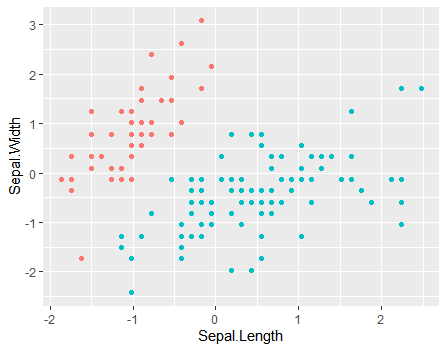
グラフ化したいけど、irisが4次元なのでできません。仕方なしに2変数だけを使ってグラフ化してみます。
なんとPCAで次元を圧縮して、なおかつグループを書いてくれる便利メソッドがfactoextraパッケージに存在します。
library(factoextra)
fviz_cluster(kmeans2, data = myiris)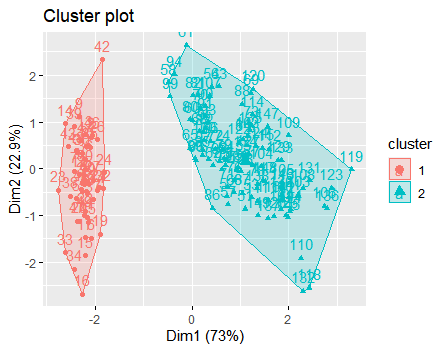
ところで、k = 2にしたけどk=3、k=4、k=5にしたときにどうなるのでしょうか。kはいったいいくつがいいのでしょうか。とりあえずプロットしてみます。
kmeans2 <-kmeans(myiris,
centers = 2,
nstart = 50)
kmeans3 <-kmeans(myiris,
centers = 3,
nstart = 50)
kmeans4 <-kmeans(myiris,
centers = 4,
nstart = 50)
kmeans5 <-kmeans(myiris,
centers = 5,
nstart = 50)
library(patchwork)
g2 <- fviz_cluster(kmeans2, data = myiris)
g3 <- fviz_cluster(kmeans3, data = myiris)
g4 <- fviz_cluster(kmeans4, data = myiris)
g5 <- fviz_cluster(kmeans5, data = myiris)
(g2+g3)/(g4+g5)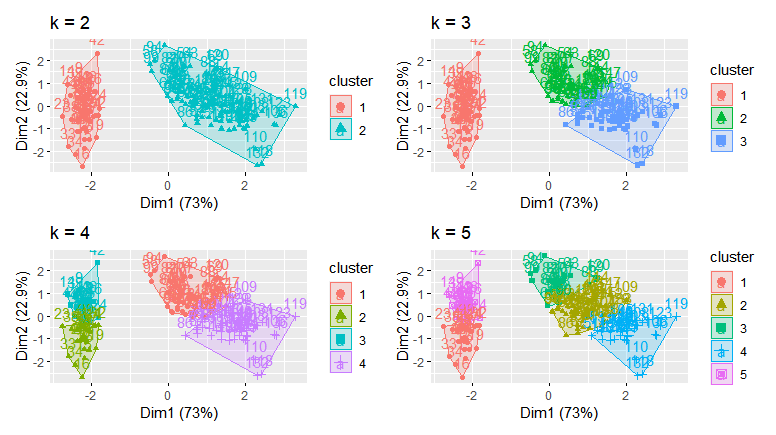
視覚的には、k=2のときが一番よさそうです。定量的にkの数を決めたいです。
定量的にkの数を決める
定量的にクラスターの評価をするには、以下の方法があります。
- Elbow method
- Silhouette method
- Gap statistic
Elbow method
肘メソッドは、within-cluster sum of square the compactness of the clusteringというものを指標にしています。kmeansの返り値のtot.withinssという値です。kmeans2$tot.withinssで取り出せる数値です。
以下のコードで解析・プロットできます。
factoextra::fviz_nbclust(myiris,
FUNcluster = kmeans,
method = "wss")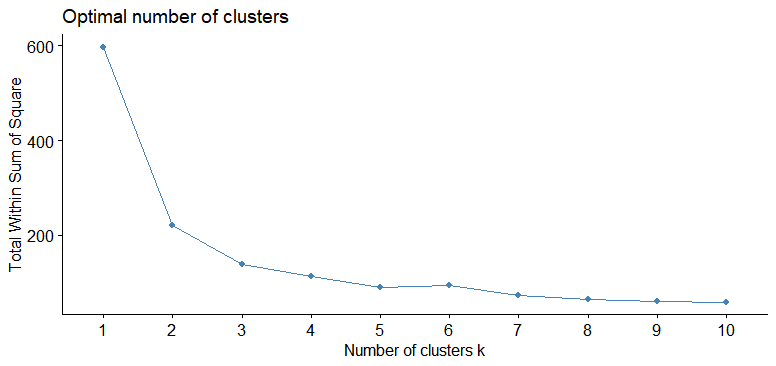
肘の曲がったところが、いいところです。つまり、2ですね!
Silhouette method
出来上がったクラスターのシルエットスコアというものを計算します。先程のmethodをsilhouetteに帰るだけです。シルエットのスペリングを95%間違えますのでお気を付けを。
factoextra::fviz_nbclust(myiris,
FUNcluster = kmeans,
method = "silhouette")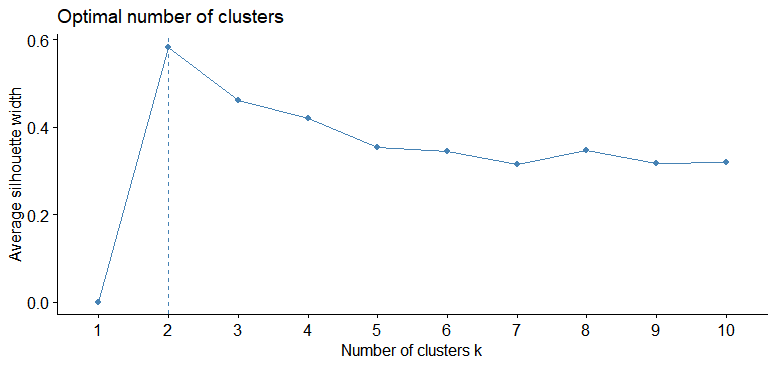
これは、シルエットの美しさポイントが一番高いところが選択ポイントになります。k=2です!
Gap statistic
ギャップ統計値というのを計算します。
factoextra::fviz_nbclust(myiris,
FUNcluster = kmeans,
method = "gap_stat")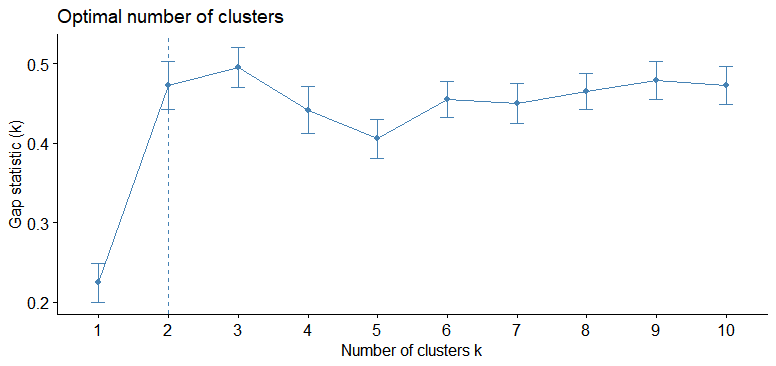
K=2のようです。初めは、あれ?k=3じゃないのと思いました。この方法は、現在のKのgap statisticが、次のKのgap statisticの1標準偏差内となる最小のKを選択するようです。
どの方法も、K=2がいいと示しました!(ほんとは、K=3が正解。。)
