アンチバイオグラム作成ツールを開発しました。
アンチバイオグラム作成ツールのダウンロードは以下のリンクから。
動機
アンチバイオグラムは、データの処理がとても大変です。さらに、重複処理はプログラミングの技術が必要になります。忙しい臨床の先生だと作ろうという気さえ起こらないでしょう。
JANISに参加している病院はアンチバイオグラム作成ツールをダウンロードできるみたいですが、参加していない病院は使えないですよね。その上、動物病院はJANISに参加できないので、入手可能なアンチバイオグラム作成ソフトはかなり限られていると思います。医者の先生も獣医の先生もぜひ使ってください。
そこで自分で作ってしまえと思って作りました。作り方は、アンチバイオグラム作成ガイドライン 2019 年 3 月 感染症教育コンソーシアム アンチバイオグラム作成ガイドライン 作成チーム を参考にしました。
セールスポイント
開発したこのツールのセールスポイントは、なんていっても簡単なところです。2クリックでアンチバイオグラムが作れます。もう一つは、複数の重複処理を試すことができるところです。重複処理は様々な方法が提案されていますが、他のアンチバイオグラム作成ソフトだと1種類の処理しかできないと思います。。。といっても使ったことないからわからないです、すいません。あと、エクセルだけで完結させたというところは努力したポイントです。サンプルデータは架空の患者です。
動作の前提条件
・Windows10で、Microsoft 365のExcelで開発・動作確認しました。
・Microsoft Excelのマクロが使用できる環境が必要です。
・薬剤感受性試験データを、当エクセルファイルに貼り付けできることが必要です。
・薬剤感受性試験のそれぞれのレコードが、①カルテ番号、②採取日、③菌名、④薬剤に対するS(Susceptible)I(Intermediate)R(Resistant )の情報を有していることが必要です。
計算方法
感受性割合 = Sの数 / ( Sの数 + Iの数 + Rの数 )
重複処理アルゴリズム
重複処理は、同一患者において同一の菌種が検出された場合に実施します。目的は、バイアスを減らすことです。当ツールでは以下3通りの重複処理方法を実施できます。
・重複処理アルゴリズムA ー 患者あたり1株を採用。初回分離株、複数検出された場合はもっとも多剤耐性の株を採用。
・重複処理アルゴリズムB ー 期間あたり1株を採用。初回分離株を採用し、その後 30 日以内は採用しない。
・重複処理アルゴリズムC ー 表現型あたり1株を採用。これまでの採用株に比べ、SIRパターンが異なる場合には採用。
使い方
1.zipファイルをダウンロードし、解凍してファイルを開いてください。Microsoft Excelのセキュリティの警告でマクロが無効化される場合には、コンテンツの有効化をしてください。
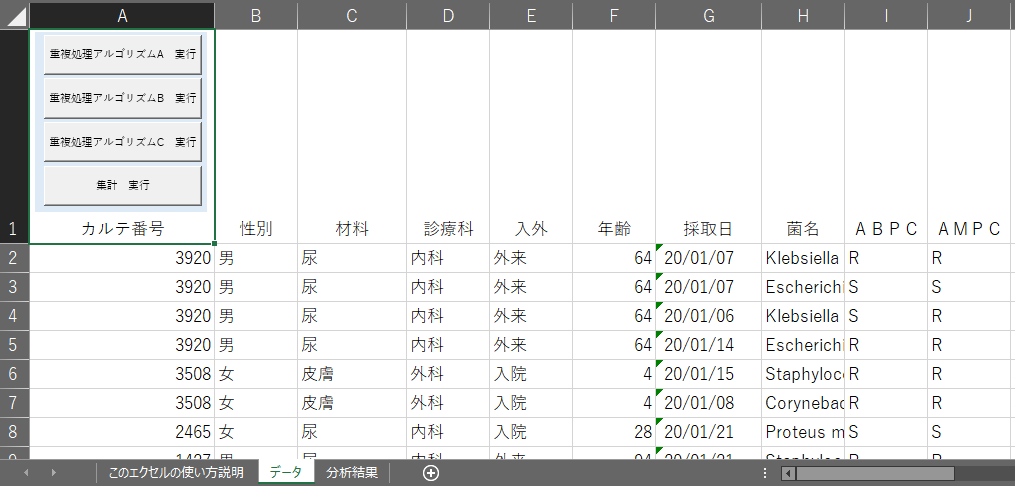
2.シート名「データ」に、ご利用者がお持ちの薬剤感受性試験データを貼り付けてください。貼り付ける前にサンプルデータは全て消してください。サンプルデータの列の並びに合わせて、患者データを加工する必要はありません。データの列の順番は問いません。列数は増えても問題ありません。
カルテ番号、採取日、菌名、SIRの情報は必要です。感受性の判定は、SかRかIの3種類で記載してください。
3.シート名「データ」の左上にある、ボタン「重複処理アルゴリズムA 実行」「重複処理アルゴリズムB 実行」「重複処理アルゴリズムC 実行」のいずれかを押してください。実行すると、一番右端の列に結果が追加されていきます。1つ実行した後に、他のアルゴリズムも実行できます。
・カルテ番号の列を尋ねる画面がでてきます。列の名前をアルファベットの半角で入力してください。
・検体採取日付の列を尋ねる画面がでてきます。列の名前をアルファベットの半角で入力してください。
・菌名の列を尋ねる画面がでてきます。列の名前をアルファベットの半角で入力してください。
・感受性判定列の範囲を尋ねる画面がでてきます。左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。
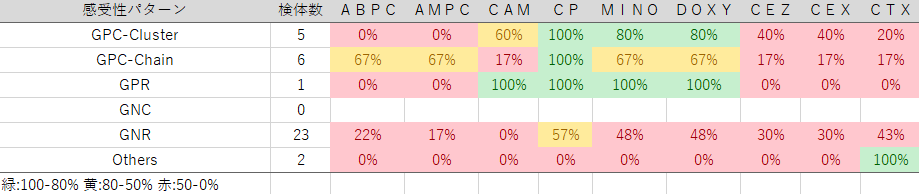
4.シート名「データ」の左上にある、ボタン「集計実行」を押してください。シート名「分析結果」に結果が表示されます。
・集計に使用する重複処理判定列を尋ねる画面がでてきます。重複処理判定列とは、さきほどの重複処理アルゴリズム実行で、一番右に追加された列のことです。列の名前をアルファベットの半角で入力してください。
・菌名の列を尋ねる画面がでてきます。列の名前をアルファベットの半角で入力してください。
・感受性判定列の範囲を尋ねる画面がでてきます。左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。
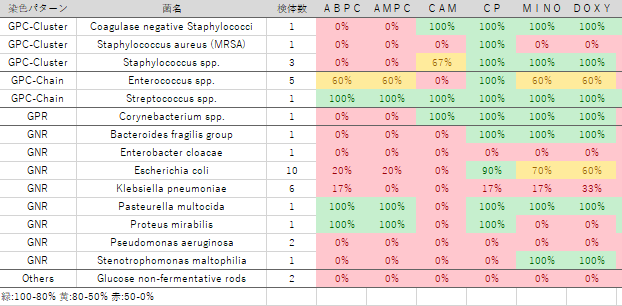
5. シート名「分析結果」を確認してください。菌が正しくグラム染色パターンに分類されていない場合は、シート名「グラム染色パターン表」で、分類に追加したい菌種を追加してください。
プライバシーポリシー
利用者の個人情報及び患者情報の収集は一切行いません。
免責事項
1.当ファイルをご利用する前に免責事項に同意し、同意後にご利用を開始してください。当ファイルをダウンロードすることによってご利用者は、免責事項に同意したものとみなします。
2.当ファイルのご利用者は、自らの行為に一切の責任を負うものとし、当ファイルによって生じた問題については、ご利用者自らで解決し、製作者は一切免責されるものとします。
3.製作者は、ご利用者のファイルの利用により発生した、ご利用者の損害又は第三者の損害に対し、いかなる責任も負いません。製作者は、当ファイル使用によるコンピューターシステムの破損、データ破損、得られたデータの不正確性、及びそれ以外のいかなる原因に基づき生じた損害について賠償する義務を一切負わないものとします。
4.本プログラムは、医薬品、医療機器等の品質、有効性及び安全性の確保等に関する法律の承認を受けたものではありません。そのため、診療には使用しないでください。個人的な研究又は娯楽の用途でお使いください。
個人利用・商用利用 可能
再配布 可能
改善点
グラム染色パターンに対応していないので、時間があるときに修正したいと思います。(→ ver1.3から対応しました)もし、以下のソースコードでアルゴリズムが間違っていることを発見した場合には、教えて頂ければありがたいです。
ソースコード
もしセキュリティ上の問題からダウンロードをしたくない、できないという方がいたら以下の方法で使用できます。
エクセルを開き、開発というタブからVisual Basicを選択してください。
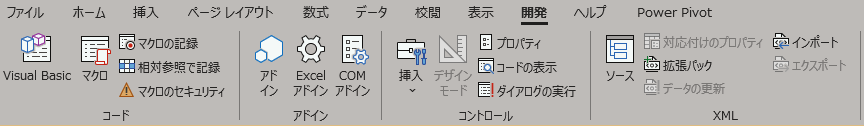
もし、開発というタブがなければ、ファイル → その他 → オプション → リボンのユーザー設定 → 開発にチェックをいれてください
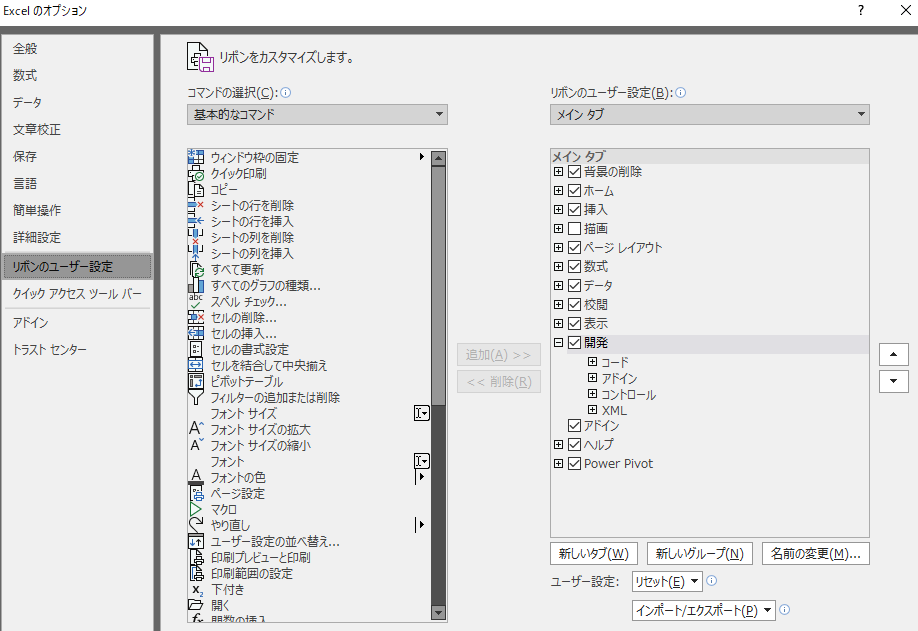
Visual Basicが開くと、以下のような画面になります。あとから記載するコードを右下のように貼り付けてください。sub ○○() / end sub で区切られたところにマウスカーソルを置いておき、 再生ボタン(緑の矢頭)を押すと、そのアルゴリズムが実行できます。
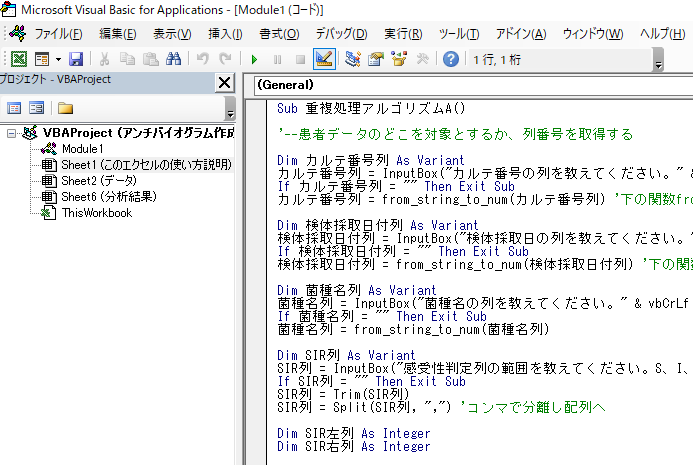
ソースコードは以下です。ver1.3
Sub 重複処理アルゴリズムA()
'--患者データのどこを対象とするか、列番号を取得する
Dim カルテ番号列 As Variant
カルテ番号列 = InputBox("カルテ番号の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:a(A列のこと)")
If カルテ番号列 = "" Then Exit Sub
カルテ番号列 = from_string_to_num(カルテ番号列) '下の関数from_string_to_numを使用
Dim 検体採取日付列 As Variant
検体採取日付列 = InputBox("検体採取日の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:g(G列のこと)")
If 検体採取日付列 = "" Then Exit Sub
検体採取日付列 = from_string_to_num(検体採取日付列) '下の関数from_string_to_numを使用
Dim 菌種名列 As Variant
菌種名列 = InputBox("菌種名の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:h(H列のこと)")
If 菌種名列 = "" Then Exit Sub
菌種名列 = from_string_to_num(菌種名列)
Dim SIR列 As Variant
SIR列 = InputBox("感受性判定列の範囲を教えてください。S、I、Rで判定されていることが前提です。" & vbCrLf & "左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:i,al( I列が左端で、AL列が右端 )")
If SIR列 = "" Then Exit Sub
SIR列 = Trim(SIR列)
SIR列 = Split(SIR列, ",") 'コンマで分離し配列へ
Dim SIR左列 As Integer
Dim SIR右列 As Integer
SIR左列 = from_string_to_num(SIR列(0)) '下の関数from_string_to_numを使用
SIR右列 = from_string_to_num(SIR列(1)) '下の関数from_string_to_numを使用
'--患者データシートの配列入手
Dim データ
Dim Lrow1 As Integer
Dim Lcol1 As Integer
Lrow1 = Worksheets("データ").Range("A1").End(xlDown).Row
Lcol1 = Worksheets("データ").Range("A1").End(xlToRight).Column
データ = Worksheets("データ").Range(Cells(2, 1), Cells(Lrow1, Lcol1)).Value '配列に代入
'以下のifアルゴリズムの結果を格納する判定配列を作成する
Dim 重複処理アルゴリズムA判定配列() As Variant '配列の要素数が動的変数なための工夫1
ReDim 重複処理アルゴリズムA判定配列(Lrow1 - 2) As Variant '配列の要素数が動的変数なための工夫2
Dim カルテ番号 As Variant
Dim 検体採取日付 As Date
Dim 菌種名 As String
Dim i As Integer
Dim j As Integer
Dim k As Integer
Dim 耐性数I As Integer
Dim 耐性数J As Integer
For i = 1 To Lrow1 - 1 '患者データを順番に処理していく
カルテ番号 = データ(i, カルテ番号列)
検体採取日付 = データ(i, 検体採取日付列)
菌種名 = データ(i, 菌種名列)
For j = 1 To Lrow1 - 1
If Not i = j Then 'if_1 現レコードとチェックする他レコードが違う場合のみ以降を実行
If カルテ番号 = データ(j, カルテ番号列) Then 'if_1_1 もし他のレコードに同じカルテ番号があった場合
If 菌種名 = データ(j, 菌種名列) Then 'if_1_1_1 もし他のレコードが同じ菌種だった場合
If 検体採取日付 > データ(j, 検体採取日付列) Then 'if_1_1_1_1 もし他のレコードが、前の日付である場合は、現在のレコードを棄却
重複処理アルゴリズムA判定配列(i - 1) = "×" 'if_1_1_1_1 判定配列に×を入れる
ElseIf 検体採取日付 = データ(j, 検体採取日付列) Then 'if_1_1_1_2 もし他のレコードが、同じ日付である場合には、最も多剤耐性の株を選ぶ
耐性数I = 0 'if_1_1_1_2 耐性数のカウントを実施。
耐性数J = 0
For k = SIR左列 To SIR右列
If データ(i, k) = "R" Then
耐性数I = 耐性数I + 1
End If
If データ(j, k) = "R" Then
耐性数J = 耐性数J + 1
End If
Next k
If 耐性数I < 耐性数J Then 'if_1_1_1_2_1 耐性数のカウントを比較。他のレコードに比べ耐性数が少なければ、現在のレコードを棄却。
重複処理アルゴリズムA判定配列(i - 1) = "×" 'if_1_1_1_2_1 判定配列に×を入れる。
ElseIf 耐性数I = 耐性数J Then 'if_1_1_1_2_2 耐性数のカウントを比較。同じ場合は、番号が小さい行を採用することにする!
If i > j Then 'if_1_1_1_2_2_1 現レコードほうが行数が大きい場合
重複処理アルゴリズムA判定配列(i - 1) = "×" 'if_1_1_1_2_2_1 判定配列に×を入れる
End If
End If
End If
End If
End If
End If
Next j
Next i
On Error GoTo error
Worksheets("データ").Range(Cells(2, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).Value = WorksheetFunction.Transpose(重複処理アルゴリズムA判定配列) '判定配列書き出し
error:
Worksheets("データ").Cells(1, Lcol1 + 1).Value = "重複処理アルゴリズムA判定" & vbCrLf & "棄却株は×" '新たな列の一番上に名前をつける
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).HorizontalAlignment = xlCenter '新たな列の一番上の中央揃え 水平
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).VerticalAlignment = xlCenter
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(1, Lcol1 + 1)).Columns.AutoFit '新たな列の文字列のサイズに列幅を合わす'新たな列の一番上の中央揃え 垂直
End Sub
Sub 重複処理アルゴリズムB()
'--患者データのどこを対象とするか、列番号を取得する
Dim カルテ番号列 As Variant
カルテ番号列 = InputBox("カルテ番号の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:a(A列のこと)")
If カルテ番号列 = "" Then Exit Sub
カルテ番号列 = from_string_to_num(カルテ番号列) '下の関数from_string_to_numを使用
Dim 検体採取日付列 As Variant
検体採取日付列 = InputBox("検体採取日の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:g(G列のこと)")
If 検体採取日付列 = "" Then Exit Sub
検体採取日付列 = from_string_to_num(検体採取日付列) '下の関数from_string_to_numを使用
Dim 検体採取日付列数字 As Integer
検体採取日付列数字 = 検体採取日付列
Dim 菌種名列 As Variant
菌種名列 = InputBox("菌種名の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:h(H列のこと)")
If 菌種名列 = "" Then Exit Sub
菌種名列 = from_string_to_num(菌種名列)
Dim SIR列 As Variant
SIR列 = InputBox("感受性判定列の範囲を教えてください。S、I、Rで判定されていることが前提です。" & vbCrLf & "左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:i,al( I列が左端で、AL列が右端 )")
If SIR列 = "" Then Exit Sub
SIR列 = Trim(SIR列)
SIR列 = Split(SIR列, ",") 'コンマで分離し配列へ
Dim SIR左列 As Integer
Dim SIR右列 As Integer
SIR左列 = from_string_to_num(SIR列(0)) '下の関数from_string_to_numを使用
SIR右列 = from_string_to_num(SIR列(1)) '下の関数from_string_to_numを使用
'--患者データシートの配列入手
Dim データ()
Dim Lrow1 As Integer
Dim Lcol1 As Integer
Lrow1 = Worksheets("データ").Range("A1").End(xlDown).Row
Lcol1 = Worksheets("データ").Range("A1").End(xlToRight).Column
データ = Worksheets("データ").Range(Cells(2, 1), Cells(Lrow1, Lcol1)).Value '配列に代入
'データを検体採取日で並び替えするが、元の順番を保存するために1列行番号を追加したデータ2を作る
Dim データ2() As Variant
ReDim データ2(1 To (Lrow1 - 1), 1 To (Lcol1 + 1)) As Variant
Dim s, t As Integer
For s = 1 To (Lrow1 - 1)
For t = 1 To Lcol1
データ2(s, t) = データ(s, t)
Next t
データ2(s, Lcol1 + 1) = s
Next s
'並び替えを行う。 arraysort(データ、開始行、終了行、基準列)
Call ArraySort(データ2(), 1, (Lrow1 - 1), 検体採取日付列数字)
'以下のifアルゴリズムの結果を格納する判定配列を作成する
Dim 重複処理アルゴリズムB判定配列() As Variant '配列の要素数が動的変数なための工夫1
ReDim 重複処理アルゴリズムB判定配列(Lrow1 - 2) As Variant '配列の要素数が動的変数なための工夫2
Dim カルテ番号 As Variant
Dim 検体採取日付 As Date
Dim 菌種名 As String
Dim 他レコード検体採取日付 As Date
Dim i As Integer
Dim j As Integer
Dim k As Integer
Dim 耐性数I As Integer
Dim 耐性数J As Integer
For i = 1 To Lrow1 - 1 '患者データを順番に処理していく
カルテ番号 = データ2(i, カルテ番号列)
検体採取日付 = データ2(i, 検体採取日付列)
菌種名 = データ2(i, 菌種名列)
For j = 1 To Lrow1 - 1
他レコード検体採取日付 = データ2(j, 検体採取日付列)
If Not i = j Then 'if_1 現レコードとチェックする他レコードが違う場合のみ以降を実行
If カルテ番号 = データ2(j, カルテ番号列) Then 'if_1_1 もし他レコードに同じカルテ番号があった場合
If 菌種名 = データ2(j, 菌種名列) Then 'if_1_1_1 もし他レコードが同じ菌種であった場合
If 検体採取日付 > 他レコード検体採取日付 Then 'if_1_1_1_1 もし他レコードが、前の日付である場合について
If (検体採取日付 - 他レコード検体採取日付) <= 30 Then 'if_1_1_1_1_1 もし他レコードが、30日以内なら
If 重複処理アルゴリズムB判定配列(j - 1) <> "×" Then 'if_1_1_1_1_1_1 もし他レコードが、採用されているなら
重複処理アルゴリズムB判定配列(i - 1) = "×" 'if_1_1_1_1_1_1 判定配列に×を入れる。
End If
End If
ElseIf 検体採取日付 = データ2(j, 検体採取日付列) Then 'if_1_1_1_2 もし他のレコードが、同じ日付である場合には、最も多剤耐性の株を選ぶ
耐性数I = 0 'if_1_1_1_2 耐性数のカウントを実施。
耐性数J = 0
For k = SIR左列 To SIR右列
If データ2(i, k) = "R" Then
耐性数I = 耐性数I + 1
End If
If データ2(j, k) = "R" Then
耐性数J = 耐性数J + 1
End If
Next k
If 耐性数I < 耐性数J Then 'if_1_1_1_2_1 耐性数のカウントを比較。他のレコードに比べ耐性数が少なければ、現在のレコードを棄却。
重複処理アルゴリズムB判定配列(i - 1) = "×" 'if_1_1_1_2_1 判定配列に×を入れる。
ElseIf 耐性数I = 耐性数J Then 'if_1_1_1_2_2 耐性数のカウントを比較。同じ場合は、番号が小さい行を採用することにする!
If i > j Then 'if_1_1_1_2_2_1 現レコードほうが行数が大きい場合
重複処理アルゴリズムB判定配列(i - 1) = "×" 'if_1_1_1_2_2_1 判定配列に×を入れる
End If
End If
End If
End If
End If
End If
Next j
Next i
' 重複処理アルゴリズムB判定配列は、検体採取日で並び替えがなされているので、行番号を使って元に戻す。
Dim 並替前_重複処理アルゴリズムB判定配列() As Variant '配列の要素数が動的変数なための工夫1
ReDim 並替前_重複処理アルゴリズムB判定配列(Lrow1 - 2) As Variant '配列の要素数が動的変数なための工夫2
Dim 行番号
Dim r As Integer
For r = 0 To UBound(重複処理アルゴリズムB判定配列)
行番号 = データ2(r + 1, (Lcol1 + 1)) 'データ2の補助列(行番号)を参照する
並替前_重複処理アルゴリズムB判定配列(行番号 - 1) = 重複処理アルゴリズムB判定配列(r)
Next r
On Error GoTo error
Worksheets("データ").Range(Cells(2, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).Value = WorksheetFunction.Transpose(並替前_重複処理アルゴリズムB判定配列) '判定配列書き出し
error:
Worksheets("データ").Cells(1, Lcol1 + 1).Value = "重複処理アルゴリズムB判定" & vbCrLf & "棄却株は×" '新たな列の一番上に名前をつける
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).HorizontalAlignment = xlCenter '新たな列の一番上の中央揃え 水平
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).VerticalAlignment = xlCenter
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(1, Lcol1 + 1)).Columns.AutoFit '新たな列の文字列のサイズに列幅を合わす'新たな列の一番上の中央揃え 垂直
End Sub
Sub 重複処理アルゴリズムC()
'--患者データのどこを対象とするか、列番号を取得する
Dim カルテ番号列 As Variant
カルテ番号列 = InputBox("カルテ番号の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:a(A列のこと)")
If カルテ番号列 = "" Then Exit Sub
カルテ番号列 = from_string_to_num(カルテ番号列) '下の関数from_string_to_numを使用
Dim 検体採取日付列 As Variant
検体採取日付列 = InputBox("検体採取日の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:g(G列のこと)")
If 検体採取日付列 = "" Then Exit Sub
検体採取日付列 = from_string_to_num(検体採取日付列) '下の関数from_string_to_numを使用
Dim 菌種名列 As Variant
菌種名列 = InputBox("菌種名の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:h(H列のこと)")
If 菌種名列 = "" Then Exit Sub
菌種名列 = from_string_to_num(菌種名列)
Dim SIR列 As Variant
SIR列 = InputBox("感受性判定列の範囲を教えてください。S、I、Rで判定されていることが前提です。" & vbCrLf & "左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:i,al( I列が左端で、AL列が右端 )")
If SIR列 = "" Then Exit Sub
SIR列 = Trim(SIR列)
SIR列 = Split(SIR列, ",") 'コンマで分離し配列へ
Dim SIR左列 As Integer
Dim SIR右列 As Integer
SIR左列 = from_string_to_num(SIR列(0)) '下の関数from_string_to_numを使用
SIR右列 = from_string_to_num(SIR列(1)) '下の関数from_string_to_numを使用
'--患者データシートの配列入手
Dim データ
Dim Lrow1 As Integer
Dim Lcol1 As Integer
Lrow1 = Worksheets("データ").Range("A1").End(xlDown).Row
Lcol1 = Worksheets("データ").Range("A1").End(xlToRight).Column
データ = Worksheets("データ").Range(Cells(2, 1), Cells(Lrow1, Lcol1)).Value '配列に代入
'SIRパターン配列を作る
Dim SIRパターン配列() As Variant
ReDim SIRパターン配列(1 To Lrow1 - 1) As Variant
Dim s As Integer
Dim t As Integer
For s = 1 To (Lrow1 - 1)
For t = SIR左列 To SIR右列
SIRパターン配列(s) = SIRパターン配列(s) + データ(s, t)
Next t
Next s
'以下のifアルゴリズムの結果を格納する判定配列を作成する
Dim 重複処理アルゴリズムC判定配列() As Variant '配列の要素数が動的変数なための工夫1
ReDim 重複処理アルゴリズムC判定配列(Lrow1 - 2) As Variant '配列の要素数が動的変数なための工夫2
Dim カルテ番号 As Variant
Dim 検体採取日付 As Date
Dim 菌種名 As String
Dim SIRパターン As String
Dim i As Integer
Dim j As Integer
Dim k As Integer
Dim 耐性数I As Integer
Dim 耐性数J As Integer
For i = 1 To Lrow1 - 1 '患者データを順番に処理していく
カルテ番号 = データ(i, カルテ番号列)
検体採取日付 = データ(i, 検体採取日付列)
菌種名 = データ(i, 菌種名列)
SIRパターン = SIRパターン配列(i)
For j = 1 To Lrow1 - 1
If Not i = j Then 'if_1 現レコードとチェックする他レコードが違う場合のみ以降を実行
If カルテ番号 = データ(j, カルテ番号列) Then 'if_1_1 もし他のレコードに同じカルテ番号があった場合
If 菌種名 = データ(j, 菌種名列) Then 'if_1_1_1 もし他のレコードが同じ菌種だった場合
If SIRパターン = SIRパターン配列(j) Then 'if_1_1_1_1 もし他のレコードが同じSIRパターンだった場合
If 検体採取日付 > データ(j, 検体採取日付列) Then 'if_1_1_1_1_1 もし他のレコードが、前の日付である場合は、現在のレコードを棄却
重複処理アルゴリズムC判定配列(i - 1) = "×" 'if_1_1_1_1_1 判定配列に×を入れる
ElseIf 検体採取日付 = データ(j, 検体採取日付列) Then 'if_1_1_1_1_2 もし他のレコードが、同じ日付である場合には、行数が大きいレコードを棄却
If i > j Then 'if_1_1_1_1_2_1 現レコードほうが行数が大きい場合
重複処理アルゴリズムC判定配列(i - 1) = "×" 'if_1_1_1_1_2_1 判定配列に×を入れる
End If
End If
End If
End If
End If
End If
Next j
Next i
On Error GoTo error
Worksheets("データ").Range(Cells(2, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).Value = WorksheetFunction.Transpose(重複処理アルゴリズムC判定配列) '判定配列書き出し
error:
Worksheets("データ").Cells(1, Lcol1 + 1).Value = "重複処理アルゴリズムC判定" & vbCrLf & "棄却株は×" '新たな列の一番上に名前をつける
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).HorizontalAlignment = xlCenter '新たな列の一番上の中央揃え 水平
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(Lrow1 - 2 + 2, Lcol1 + 1)).VerticalAlignment = xlCenter
Worksheets("データ").Range(Cells(1, Lcol1 + 1), Cells(1, Lcol1 + 1)).Columns.AutoFit '新たな列の文字列のサイズに列幅を合わす'新たな列の一番上の中央揃え 垂直
End Sub
Sub 集計()
'グラム染色パターン表から、データを取得
ThisWorkbook.Worksheets("グラム染色パターン表").Activate
'GPC_clusterの配列を取得
Dim Lrow_GPC_cluster As Integer
Lrow_GPC_cluster = Worksheets("グラム染色パターン表").Range("A1").End(xlDown).Row
Dim GPC_cluster As Variant
GPC_cluster = Worksheets("グラム染色パターン表").Range(Cells(2, 1), Cells(Lrow_GPC_cluster, 1)).Value
'GPC_chainの配列を取得
Dim Lrow_GPC_chain As Integer
Lrow_GPC_chain = Worksheets("グラム染色パターン表").Range("B1").End(xlDown).Row
Dim GPC_chain As Variant
GPC_chain = Worksheets("グラム染色パターン表").Range(Cells(2, 2), Cells(Lrow_GPC_chain, 2)).Value
'GPRの配列を取得
Dim Lrow_GPR As Integer
Lrow_GPR = Worksheets("グラム染色パターン表").Range("C1").End(xlDown).Row
Dim GPR As Variant
GPR = Worksheets("グラム染色パターン表").Range(Cells(2, 3), Cells(Lrow_GPR, 3)).Value
'GNCの配列を取得
Dim Lrow_GNC As Integer
Lrow_GNC = Worksheets("グラム染色パターン表").Range("D1").End(xlDown).Row
Dim GNC As Variant
GNC = Worksheets("グラム染色パターン表").Range(Cells(2, 4), Cells(Lrow_GNC, 4)).Value
'GNRの配列を取得
Dim Lrow_GNR As Integer
Lrow_GNR = Worksheets("グラム染色パターン表").Range("E1").End(xlDown).Row
Dim GNR As Variant
GNR = Worksheets("グラム染色パターン表").Range(Cells(2, 5), Cells(Lrow_GNR, 5)).Value
'1文字目の大文字小文字の区別が問題となってしまうことを防ぐため、1文字目を削除した配列にする。
Dim i As Integer
For i = 1 To UBound(GPC_cluster)
GPC_cluster(i, 1) = Mid(GPC_cluster(i, 1), 2)
Next i
For i = 1 To UBound(GPC_chain)
GPC_chain(i, 1) = Mid(GPC_chain(i, 1), 2)
Next i
For i = 1 To UBound(GPR)
GPR(i, 1) = Mid(GPR(i, 1), 2)
Next i
For i = 1 To UBound(GNC)
GNC(i, 1) = Mid(GNC(i, 1), 2)
Next i
For i = 1 To UBound(GNR)
GNR(i, 1) = Mid(GNR(i, 1), 2)
Next i
'判定等に必要な列を尋ねる
ThisWorkbook.Worksheets("データ").Activate
Dim 判定列 As Variant
判定列 = InputBox("集計に使用する重複処理アルゴリズム判定列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:am(AM列のこと)")
Debug.Print
If 判定列 = "" Then Exit Sub ' キャンセル時に終了
判定列 = from_string_to_num(判定列) '下の関数from_string_to_numを使用
Dim 菌種名列 As Variant
菌種名列 = InputBox("菌種名の列を教えてください。" & vbCrLf & "アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:h(H列のこと)")
If 菌種名列 = "" Then Exit Sub
菌種名列 = from_string_to_num(菌種名列)
Dim SIR列 As Variant
SIR列 = InputBox("感受性判定列の範囲を教えてください。S、I、Rで判定されていることが前提です。" & vbCrLf & "左端の列名と右端の列名をコンマで区切り、アルファベットで記入してください。" & vbCrLf & vbCrLf & "例:i,al( I列が左端で、AL列が右端 )")
If SIR列 = "" Then Exit Sub
SIR列 = Trim(SIR列)
SIR列 = Split(SIR列, ",") 'コンマで分離し配列へ
Dim SIR左列 As Integer
Dim SIR右列 As Integer
SIR左列 = from_string_to_num(SIR列(0)) '下の関数from_string_to_numを使用
SIR右列 = from_string_to_num(SIR列(1)) '下の関数from_string_to_numを使用
'--患者データシートの配列入手
Dim Lrow1 As Integer
Dim Lcol1 As Integer
Lrow1 = Worksheets("データ").Range("A1").End(xlDown).Row
Lcol1 = Worksheets("データ").Range("A1").End(xlToRight).Column
Dim SIRデータ() As Variant 'SIRデータ入った表
SIRデータ = Worksheets("データ").Range(Cells(2, SIR左列), Cells(Lrow1, SIR右列)).Value
Dim 抗菌薬ラベル() As Variant '抗菌薬名の1行
抗菌薬ラベル = Worksheets("データ").Range(Cells(1, SIR左列), Cells(1, SIR右列)).Value
Dim 菌種名配列() As Variant '菌種名の入った1列
菌種名配列 = Worksheets("データ").Range(Cells(2, 菌種名列), Cells(Lrow1, 菌種名列)).Value
Dim 判定配列() As Variant '判定の入った1列
判定配列 = Worksheets("データ").Range(Cells(2, 判定列), Cells(Lrow1, 判定列)).Value
Dim ユニーク菌種名配列() As Variant '菌種名を重複のない1列にする
ユニーク菌種名配列 = WorksheetFunction.Unique(Worksheets("データ").Range(Cells(2, 菌種名列), Cells(Lrow1, 菌種名列)).Value)
Dim 菌の数 As Integer
菌の数 = UBound(ユニーク菌種名配列) '菌の数を数える
Dim 薬の数 As Integer
薬の数 = SIR右列 - SIR左列 + 1
Call ArraySort(ユニーク菌種名配列(), 1, 菌の数, 1) '並び替えする
Dim 検体数() As Variant '検体数を数える1列
ReDim 検体数(1 To 菌の数, 1 To 1)
Dim 感受性表_Sの数() As Variant 'Sを数える表。行は菌の数、列は薬の数
ReDim 感受性表_Sの数(1 To 菌の数, 1 To (薬の数))
Dim 感受性表_SIRの数() As Variant 'SIRを数える表。行は菌の数、列は薬の数
ReDim 感受性表_SIRの数(1 To 菌の数, 1 To (薬の数))
Dim 菌 As String
Dim s As Integer
Dim t As Integer
Dim u As Integer
For s = 1 To 菌の数 'ユニーク菌種名配列を1行づつ処理していく
菌 = ユニーク菌種名配列(s, 1)
For t = 1 To (Lrow1 - 1) 'SIRデータを1行づつ処理していく
If 菌 = 菌種名配列(t, 1) Then 'ユニーク菌種名配列と同じ菌を見つけた場合
If Not 判定配列(t, 1) = "×" Then
検体数(s, 1) = 検体数(s, 1) + 1 '見つけたら、検体数をカウントアップ
For u = 1 To (薬の数) 'SIRデータの1行を、1列づつ処理していく
If SIRデータ(t, u) = "S" Then 'SIRデータが、Sだった場合
感受性表_Sの数(s, u) = 感受性表_Sの数(s, u) + 1 'Sをカウントアップ
感受性表_SIRの数(s, u) = 感受性表_SIRの数(s, u) + 1 'SIRをカウントアップ
ElseIf SIRデータ(t, u) = "I" Or SIRデータ(t, u) = "R" Then 'SIRデータが、IもしくはRだった場合
感受性表_SIRの数(s, u) = 感受性表_SIRの数(s, u) + 1 'SIRをカウントアップ
End If
Next u
End If
End If
Next t
Next
'感受性表を作るために、S/SIRの計算をする
Dim 感受性表() As Variant 'SIRの感受性の表。行は菌の数、列は薬の数
ReDim 感受性表(1 To 菌の数, 1 To (薬の数))
Dim v As Integer
Dim w As Integer
For v = 1 To 菌の数
For w = 1 To (薬の数) ' 1行ごとに処理
If Not 感受性表_SIRの数(v, w) = 0 Then ' 分母が0の場合は除く
感受性表(v, w) = 感受性表_Sの数(v, w) / 感受性表_SIRの数(v, w) '割り算 S/SIR
End If
Next w
Next v
'結果の書き出し
ThisWorkbook.Worksheets("分析結果").Activate
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 1)).Value = "染色パターン"
Worksheets("分析結果").Range(Cells(1, 2), Cells(1, 2)).Value = "菌名"
Worksheets("分析結果").Range(Cells(1, 3), Cells(1, 3)).Value = "検体数"
Worksheets("分析結果").Range(Cells(1, 4), Cells(1, 3 + (薬の数))).Value = 抗菌薬ラベル() '書き出し
'現在のユニーク菌種名配列を1行づつチェックし、染色パターンに該当する行番号を取得する。
Dim GPC_cluster_N() As Variant
Dim GPC_chain_N() As Variant
Dim GPR_N() As Variant
Dim GNC_N() As Variant
Dim GNR_N() As Variant
Dim others_N() As Variant
' incrementをそれぞれ設定する。これはユニーク菌種配列内の、それぞれの染色パターンに合致する菌の数と一致する。
Dim inc_GPC_cluster As Integer
Dim inc_GPC_chain As Integer
Dim inc_GPR As Integer
Dim inc_GNC As Integer
Dim inc_GNR As Integer
Dim inc_others As Integer
inc_GPC_cluster = 0
inc_GPC_chain = 0
inc_GPR = 0
inc_GNC = 0
inc_GNR = 0
inc_others = 0
' ユニーク菌種名配列を1行づつ染色パターン配列と合致するかチェックし、合致したらその行番号を合致する染色パターン配列に格納する。
Dim y As Integer
For y = 1 To 菌の数
For t = 1 To UBound(GPC_cluster)
If InStr(ユニーク菌種名配列(y, 1), GPC_cluster(t, 1)) > 0 Then '部分一致したら>0となる
ReDim Preserve GPC_cluster_N(inc_GPC_cluster)
GPC_cluster_N(inc_GPC_cluster) = y 'GPC_cluster_N配列に、ユニーク配列の行番号を格納する。
inc_GPC_cluster = inc_GPC_cluster + 1 'incrementを1つカウントアップする
GoTo labelA
End If
Next t
For t = 1 To UBound(GPC_chain)
If InStr(ユニーク菌種名配列(y, 1), GPC_chain(t, 1)) > 0 Then
ReDim Preserve GPC_chain_N(inc_GPC_chain)
GPC_chain_N(inc_GPC_chain) = y
inc_GPC_chain = inc_GPC_chain + 1
GoTo labelA
End If
Next t
For t = 1 To UBound(GPR)
If InStr(ユニーク菌種名配列(y, 1), GPR(t, 1)) > 0 Then
ReDim Preserve GPR_N(inc_GPR)
GPR_N(inc_GPR) = y
inc_GPR = inc_GPR + 1
GoTo labelA
End If
Next t
For t = 1 To UBound(GNC)
If InStr(ユニーク菌種名配列(y, 1), GNC(t, 1)) > 0 Then
ReDim Preserve GNC_N(inc_GNC)
GNC_N(inc_GNC) = y
inc_GNC = inc_GNC + 1
GoTo labelA
End If
Next t
For t = 1 To UBound(GNR)
If InStr(ユニーク菌種名配列(y, 1), GNR(t, 1)) > 0 Then
ReDim Preserve GNR_N(inc_GNR)
GNR_N(inc_GNR) = y
inc_GNR = inc_GNR + 1
GoTo labelA
End If
Next t
ReDim Preserve others_N(inc_others)
others_N(inc_others) = y
inc_others = inc_others + 1
labelA:
Next y
Debug.Print
'染色パターン配列ごとに書き出しを行う。onerrorがあるのは、incrementが0のときにエラーになってしまうことを想定したもの。
On Error GoTo error1
For y = 0 To inc_GPC_cluster - 1
Worksheets("分析結果").Range(Cells(y + 2, 1), Cells(y + 2, 1)).Value = "GPC-Cluster"
Worksheets("分析結果").Range(Cells(y + 2, 2), Cells(y + 2, 2)).Value = ユニーク菌種名配列(GPC_cluster_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2, 3), Cells(y + 2, 3)).Value = 検体数(GPC_cluster_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2, 4), Cells(y + 2, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, GPC_cluster_N(y), 0)
Next y
error1:
On Error GoTo error2
For y = 0 To inc_GPC_chain - 1
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1, 1), Cells(y + 2 + inc_GPC_cluster - 1 + 1, 1)).Value = "GPC-Chain"
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1, 2), Cells(y + 2 + inc_GPC_cluster - 1 + 1, 2)).Value = ユニーク菌種名配列(GPC_chain_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1, 3), Cells(y + 2 + inc_GPC_cluster - 1 + 1, 3)).Value = 検体数(GPC_chain_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1, 4), Cells(y + 2 + inc_GPC_cluster - 1 + 1, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, GPC_chain_N(y), 0)
Next y
error2:
On Error GoTo error3
For y = 0 To inc_GPR - 1
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 1), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 1)).Value = "GPR"
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 2), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 2)).Value = ユニーク菌種名配列(GPR_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 3), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 3)).Value = 検体数(GPR_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 4), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, GPR_N(y), 0)
Next y
error3:
On Error GoTo error4
For y = 0 To inc_GNC - 1
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 1), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 1)).Value = "GNC"
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 2), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 2)).Value = ユニーク菌種名配列(GNC_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 3), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 3)).Value = 検体数(GNC_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 4), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, GNC_N(y), 0)
Next y
error4:
On Error GoTo error5
For y = 0 To inc_GNR - 1
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 1), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 1)).Value = "GNR"
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 2), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 2)).Value = ユニーク菌種名配列(GNR_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 3), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 3)).Value = 検体数(GNR_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 4), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, GNR_N(y), 0)
Next y
error5:
On Error GoTo error6
For y = 0 To inc_others - 1
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 1), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 1)).Value = "Others"
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 2), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 2)).Value = ユニーク菌種名配列(others_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 3), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 3)).Value = 検体数(others_N(y), 1)
Worksheets("分析結果").Range(Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 4), Cells(y + 2 + inc_GPC_cluster - 1 + 1 + inc_GPC_chain - 1 + 1 + inc_GPR - 1 + 1 + inc_GNC - 1 + 1 + inc_GNR - 1 + 1, 3 + (薬の数))).Value = Application.WorksheetFunction.Index(感受性表, others_N(y), 0)
Next y
error6:
Worksheets("分析結果").Range(Cells(2, 4), Cells(菌の数 + 1, 3 + (薬の数))).NumberFormatLocal = "0%" 'パーセント表示にする
Worksheets("分析結果").Range(Cells(1, 1), Cells(菌の数 + 1, 3 + (薬の数))).HorizontalAlignment = xlCenter '新たな列の一番上の中央揃え 水平
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 3 + (薬の数))).Interior.Color = RGB(217, 217, 217) '列ラベルの色
Worksheets("分析結果").Cells(菌の数 + 2, 1).Value = "緑:100-80% 黄:80-50% 赤:50-0%" '色の説明追加
With Worksheets("分析結果").Range(Cells(2, 4), Cells(菌の数 + 1, 3 + (薬の数))) '条件付き書式 緑:100-80% 黄:80-50% 赤:50-0%
.FormatConditions.Delete
.FormatConditions.Add Type:=xlCellValue, Operator:=xlEqual, Formula1:="=""""" '空白のとき
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="50%"
.FormatConditions(2).Interior.Color = RGB(255, 199, 206)
.FormatConditions(2).Font.Color = RGB(156, 0, 6)
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="80%"
.FormatConditions(3).Interior.Color = RGB(255, 235, 156)
.FormatConditions(3).Font.Color = RGB(156, 101, 0)
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="101%"
.FormatConditions(4).Interior.Color = RGB(198, 239, 206)
.FormatConditions(4).Font.Color = RGB(0, 97, 0)
End With
'線の追加
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 3 + (薬の数))).Borders(xlEdgeTop).Weight = xlThin
Worksheets("分析結果").Range(Cells(1, 1), Cells(1, 3 + (薬の数))).Borders(xlEdgeTop).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + 1, 1), Cells(inc_GPC_cluster + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + 1, 1), Cells(inc_GPC_cluster + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + inc_GNR + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + inc_GNR + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + inc_GNR + 1, 1), Cells(inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + inc_GNR + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(菌の数 + 1, 1), Cells(菌の数 + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(菌の数 + 1, 1), Cells(菌の数 + 1, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
'それぞれの染色パターンごとのアンチバイオグラムを作成
Dim 感受性表_GPC_cluster() As Variant
ReDim 感受性表_GPC_cluster(0, 1 To (薬の数))
Dim 感受性表_GPC_chain() As Variant
ReDim 感受性表_GPC_chain(0, 1 To (薬の数))
Dim 感受性表_GPR() As Variant
ReDim 感受性表_GPR(0, 1 To (薬の数))
Dim 感受性表_GNC() As Variant
ReDim 感受性表_GNC(0, 1 To (薬の数))
Dim 感受性表_GNR() As Variant
ReDim 感受性表_GNR(0, 1 To (薬の数))
Dim 感受性表_others() As Variant
ReDim 感受性表_others(0, 1 To (薬の数))
Dim ii As Integer
Dim jj As Integer
Dim kk As Integer
Dim Sの数 As Integer
Dim SIRの数 As Integer
Dim 検体の数 As Integer
If Not inc_GPC_cluster = 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GPC_cluster - 1
Sの数 = Sの数 + 感受性表_Sの数(GPC_cluster_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GPC_cluster_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GPC_cluster(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GPC_cluster(0, jj) = ""
Next jj
End If
If inc_GPC_chain > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GPC_chain - 1
Sの数 = Sの数 + 感受性表_Sの数(GPC_chain_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GPC_chain_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GPC_chain(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GPC_chain(0, jj) = ""
Next jj
End If
If inc_GPC_cluster > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GPC_cluster - 1
Sの数 = Sの数 + 感受性表_Sの数(GPC_cluster_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GPC_cluster_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GPC_cluster(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GPC_cluster(0, jj) = ""
Next jj
End If
If inc_GPR > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GPR - 1
Sの数 = Sの数 + 感受性表_Sの数(GPR_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GPR_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GPR(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GPR(0, jj) = ""
Next jj
End If
If inc_GNC > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GNC - 1
Sの数 = Sの数 + 感受性表_Sの数(GNC_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GNC_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GNC(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GNC(0, jj) = ""
Next jj
End If
If inc_GNR > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_GNR - 1
Sの数 = Sの数 + 感受性表_Sの数(GNR_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(GNR_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_GNR(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_GNR(0, jj) = ""
Next jj
End If
If inc_others > 0 Then
For jj = 1 To (薬の数) ' 1行ごとに処理
Sの数 = 0
SIRの数 = 0
For ii = 0 To inc_others - 1
Sの数 = Sの数 + 感受性表_Sの数(others_N(ii), jj)
SIRの数 = SIRの数 + 感受性表_SIRの数(others_N(ii), jj)
If Not SIRの数 = 0 Then ' 分母が0の場合は除く
感受性表_others(0, jj) = Sの数 / SIRの数 '割り算 S/SIR
End If
Next ii
Next jj
Else:
For jj = 1 To (薬の数) ' 1行ごとに処理
感受性表_others(0, jj) = ""
Next jj
End If
'染色パターンごとに検体数を数える
Dim 検体数_GPC_cluster As Integer
Dim 検体数_GPC_chain As Integer
Dim 検体数_GPR As Integer
Dim 検体数_GNC As Integer
Dim 検体数_GNR As Integer
Dim 検体数_others As Integer
検体数_GPC_cluster = 0
If inc_GPC_cluster > 0 Then
For ii = 0 To inc_GPC_cluster - 1
検体数_GPC_cluster = 検体数_GPC_cluster + 検体数(GPC_cluster_N(ii), 1)
Next ii
End If
検体数_GPC_chain = 0
If inc_GPC_chain > 0 Then
For ii = 0 To inc_GPC_chain - 1
検体数_GPC_chain = 検体数_GPC_chain + 検体数(GPC_chain_N(ii), 1)
Next ii
End If
検体数_GPR = 0
If inc_GPR > 0 Then
For ii = 0 To inc_GPR - 1
検体数_GPR = 検体数_GPR + 検体数(GPR_N(ii), 1)
Next ii
End If
検体数_GNC = 0
If inc_GNC > 0 Then
For ii = 0 To inc_GNC - 1
検体数_GNC = 検体数_GNC + 検体数(GNC_N(ii), 1)
Next ii
End If
検体数_GNR = 0
If inc_GNR > 0 Then
For ii = 0 To inc_GNR - 1
検体数_GNR = 検体数_GNR + 検体数(GNR_N(ii), 1)
Next ii
End If
検体数_others = 0
If inc_others > 0 Then
For ii = 0 To inc_others - 1
検体数_others = 検体数_others + 検体数(others_N(ii), 1)
Next ii
End If
'結果の書き出し
Dim 二段目開始行 As Integer
二段目開始行 = 4 + inc_GPC_cluster + inc_GPC_chain + inc_GPR + inc_GNC + inc_GNR + inc_others
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 2)).Value = "感受性パターン"
Worksheets("分析結果").Range(Cells(1 + 二段目開始行, 2), Cells(1 + 二段目開始行, 2)).Value = "GPC-Cluster"
Worksheets("分析結果").Range(Cells(2 + 二段目開始行, 2), Cells(2 + 二段目開始行, 2)).Value = "GPC-Chain"
Worksheets("分析結果").Range(Cells(3 + 二段目開始行, 2), Cells(3 + 二段目開始行, 2)).Value = "GPR"
Worksheets("分析結果").Range(Cells(4 + 二段目開始行, 2), Cells(4 + 二段目開始行, 2)).Value = "GNC"
Worksheets("分析結果").Range(Cells(5 + 二段目開始行, 2), Cells(5 + 二段目開始行, 2)).Value = "GNR"
Worksheets("分析結果").Range(Cells(6 + 二段目開始行, 2), Cells(6 + 二段目開始行, 2)).Value = "Others"
Worksheets("分析結果").Range(Cells(二段目開始行, 3), Cells(二段目開始行, 3)).Value = "検体数"
Worksheets("分析結果").Range(Cells(1 + 二段目開始行, 3), Cells(1 + 二段目開始行, 3)).Value = 検体数_GPC_cluster
Worksheets("分析結果").Range(Cells(2 + 二段目開始行, 3), Cells(2 + 二段目開始行, 3)).Value = 検体数_GPC_chain
Worksheets("分析結果").Range(Cells(3 + 二段目開始行, 3), Cells(3 + 二段目開始行, 3)).Value = 検体数_GPR
Worksheets("分析結果").Range(Cells(4 + 二段目開始行, 3), Cells(4 + 二段目開始行, 3)).Value = 検体数_GNC
Worksheets("分析結果").Range(Cells(5 + 二段目開始行, 3), Cells(5 + 二段目開始行, 3)).Value = 検体数_GNR
Worksheets("分析結果").Range(Cells(6 + 二段目開始行, 3), Cells(6 + 二段目開始行, 3)).Value = 検体数_others
Worksheets("分析結果").Range(Cells(二段目開始行, 4), Cells(二段目開始行, 3 + (薬の数))).Value = 抗菌薬ラベル()
Worksheets("分析結果").Range(Cells(1 + 二段目開始行, 4), Cells(1 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_GPC_cluster(), 0)
Worksheets("分析結果").Range(Cells(2 + 二段目開始行, 4), Cells(2 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_GPC_chain(), 0)
Worksheets("分析結果").Range(Cells(3 + 二段目開始行, 4), Cells(3 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_GPR(), 0)
Worksheets("分析結果").Range(Cells(4 + 二段目開始行, 4), Cells(4 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_GNC(), 0)
Worksheets("分析結果").Range(Cells(5 + 二段目開始行, 4), Cells(5 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_GNR(), 0)
Worksheets("分析結果").Range(Cells(6 + 二段目開始行, 4), Cells(6 + 二段目開始行, 3 + 薬の数)).Value = Application.WorksheetFunction.Index(感受性表_others(), 0)
Worksheets("分析結果").Range(Cells(1 + 二段目開始行, 4), Cells(6 + 二段目開始行, 3 + 薬の数)).NumberFormatLocal = "0%" 'パーセント表示にする
Worksheets("分析結果").Range(Cells(二段目開始行, 1), Cells(二段目開始行, 3 + 薬の数)).HorizontalAlignment = xlCenter '水平
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(6 + 二段目開始行, 2)).HorizontalAlignment = xlCenter
Worksheets("分析結果").Range(Cells(二段目開始行, 3), Cells(6 + 二段目開始行, 3)).HorizontalAlignment = xlCenter
Worksheets("分析結果").Range(Cells(二段目開始行, 4), Cells(6 + 二段目開始行, 3 + 薬の数)).HorizontalAlignment = xlCenter
Worksheets("分析結果").Range(Cells(1, 1), Cells(二段目開始行 + 6, 3 + 薬の数)).Columns.AutoFit '列幅を調整
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 3 + (薬の数))).Interior.Color = RGB(217, 217, 217) '列ラベルの色
Worksheets("分析結果").Cells(7 + 二段目開始行, 2).Value = "緑:100-80% 黄:80-50% 赤:50-0%" '色の説明追加
With Worksheets("分析結果").Range(Cells(1 + 二段目開始行, 4), Cells(6 + 二段目開始行, 3 + (薬の数))) '条件付き書式 緑:100-80% 黄:80-50% 赤:50-0%
.FormatConditions.Delete
.FormatConditions.Add Type:=xlCellValue, Operator:=xlEqual, Formula1:="=""""" '空白のとき
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="50%"
.FormatConditions(2).Interior.Color = RGB(255, 199, 206)
.FormatConditions(2).Font.Color = RGB(156, 0, 6)
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="80%"
.FormatConditions(3).Interior.Color = RGB(255, 235, 156)
.FormatConditions(3).Font.Color = RGB(156, 101, 0)
.FormatConditions.Add Type:=xlCellValue, Operator:=xlLess, Formula1:="101%"
.FormatConditions(4).Interior.Color = RGB(198, 239, 206)
.FormatConditions(4).Font.Color = RGB(0, 97, 0)
End With
'線の追加
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 3 + (薬の数))).Borders(xlEdgeTop).Weight = xlThin
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 3 + (薬の数))).Borders(xlEdgeTop).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(二段目開始行, 2), Cells(二段目開始行, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Worksheets("分析結果").Range(Cells(6 + 二段目開始行, 2), Cells(6 + 二段目開始行, 3 + (薬の数))).Borders(xlEdgeBottom).Weight = xlThin
Worksheets("分析結果").Range(Cells(6 + 二段目開始行, 2), Cells(6 + 二段目開始行, 3 + (薬の数))).Borders(xlEdgeBottom).Color = RGB(100, 100, 100)
Debug.Print
End Sub
Function from_string_to_num(va As Variant) As Variant '変換する関数
from_string_to_num = Range(va & "1").Column '列番号を取得
End Function
Public Sub ArraySort(ByRef data() As Variant, Min As Variant, Max As Variant, key As Integer) '並替。変数(データ,開始行,終了行,並替基準列)
Dim i As Double
Dim j As Double
Dim k As Double
Dim r As Variant
Dim s As Variant
r = data(Int((Min + Max) / 2), key)
i = Min
j = Max
Do
Do While data(i, key) < r
i = i + 1
Loop
Do While data(j, key) > r
j = j - 1
Loop
If i >= j Then Exit Do
For k = LBound(data, 2) To UBound(data, 2)
s = data(i, k)
data(i, k) = data(j, k)
data(j, k) = s
Next
i = i + 1
j = j - 1
Loop
If (Min < i - 1) Then
Call ArraySort(data, Min, i - 1, key)
End If
If (Max > j + 1) Then
Call ArraySort(data, j + 1, Max, key)
End If
End Sub