DICOMをプログラミングで操作したいと思います。うちの犬のCTデータを使います。以下の項目を含んだチュートリアルになっております。
- 通常のCTの断面像の表示方法
- 3次元画像処理で多断面再構築法(MPR: Multi Planar Reconstruction)を表示する方法
- スライスのスライダーを追加する方法(matplotlib, plotly)
- マウスホバーでCT値をインタラクティブに知る方法(plotly)
なお、概要としてはDICOMからピクセルデータを取り出してnumpy arrayに変換します。numpy arrayになってしまえば、スライスの仕方によって、通常のCTの断面像とMPRを表現することができます。
実行環境はAnacondaのjupyter notebookです。DICOMを読み込むためのライブラリはpydicomを使います。pip install pydicomでインストールできます。
まず以下のコードで、試しました。失敗です。解説は、コード中にコメントで書いています。
import pydicom
import numpy as np
import matplotlib.pyplot as plt
import sys
import glob
# DICOM ファイルを読み込み
files = []
print('DICOMファイルの場所: {}'.format("C:\\Users\\patient_name/*"))
for fname in glob.glob("C:\\Users\\patient_name/*", recursive=False):
files.append(pydicom.dcmread(fname))
# slicesというリストを作成。ファイルに、slicelocationという属性があれば追加していく。
slices = []
skipcount = 0
for f in files:
if hasattr(f, 'SliceLocation'):
slices.append(f)
else:
skipcount = skipcount + 1
print("該当ファイル数: {}".format(len(slices)))
print("スライス位置がないファイル: {}".format(skipcount))
# スライスの順番をそろえる
slices = sorted(slices, key=lambda s: s.SliceLocation) # .SliceLocationで場所情報を取得
# len(slices) = 270
# アスペクト比を計算する
ps = slices[0].PixelSpacing # ps = [0.571, 0.571] 1ピクセルの [y, x] 長さ
ss = slices[0].SliceThickness # ss = "3.0" スライスの厚み
ax_aspect = ps[0]/ps[1] # yの長さ/xの長さ = 1
sag_aspect = ss/ps[0] # スライスの厚み/y軸方向への1ピクセルの長さ = 3/0.571 = 5.25
cor_aspect = ss/ps[1] # スライスの厚み/x軸方向への1ピクセルの長さ = 3/0.571 = 5.25
# 空の3Dのnumpy arrayを作成する
img_shape = list(slices[0].pixel_array.shape) # img_shape = [512, 512]
img_shape.insert(0,len(slices)) # img_shape = [270, 512, 512]
img3d = np.zeros(img_shape) # 空のarrayを作る np.zeros([270, 512, 512])
# 3Dのnumpy arrayを作る
for i, s in enumerate(slices):
img2d = s.pixel_array # img2d.shape = (512, 512)
img3d[i,:, :] = img2d # img3d.shape = (270, 512, 512)
# プロットする
middle0 = img_shape[0]//2 #3d array の(z軸)頭尾軸中央 270/2 = 135
middle1 = img_shape[1]//2 #3d array の(y軸)上下軸中央 512/2 = 256
middle2 = img_shape[2]//2 #3d array の(x軸)左右軸中央 512/2 = 256
a1 = plt.subplot(1, 3, 1)
plt.imshow(img3d[middle0,:, :])
a1.set_aspect(ax_aspect) # ax_aspect = 1
a2 = plt.subplot(1, 3, 2)
plt.imshow(img3d[:, middle1, :])
a2.set_aspect(cor_aspect) # cor_aspect = 5.25
a3 = plt.subplot(1, 3, 3)
plt.imshow(img3d[:, :, middle2]) #
a3.set_aspect(sag_aspect) # sag_aspect = 5.25
plt.show()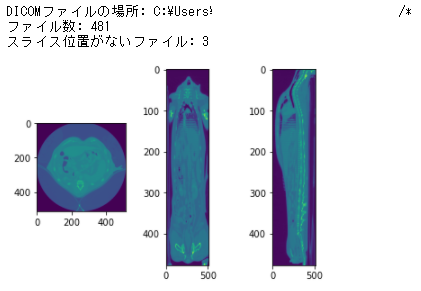
コロナルとサジタルを見ると、肝臓のところに変な線が入っていることがわかります。この原因は、CTを造影剤を入れて肝臓を複数回撮影したことによります。複数のシリーズが混ぜこぜになって同時に表示されています。CTのシリーズを特定して読み込む必要があることがわかりました。
ちなみに、断面の呼び方は以下の通りです。
File: Human anatomy planes.svg (Jul. 8, 2012). In Wikipedia: The Free Encyclopedia. Retrieved from https://commons.wikimedia.org/wiki/File:Human_anatomy_planes-JA.svg#file
以下は、上記コードを改善してシリーズを特定し読み込んでいます。18行目に1行コードを追加しています。
import pydicom
import numpy as np
import matplotlib.pyplot as plt
import sys
import glob
# DICOM ファイルを読み込み
files = []
print('DICOMファイルの場所: {}'.format("C:\\Users\\patient_name/*"))
for fname in glob.glob("C:\\Users\\patient_name/*", recursive=False):
files.append(pydicom.dcmread(fname))
# slicesというリストを作成。ファイルに、slicelocationという属性があれば追加していく。
slices = []
skipcount = 0
for f in files:
if hasattr(f, 'SliceLocation'):
if f.SeriesNumber==5: #5回CTを撮影し、5回目のデータのみを抽出
slices.append(f)
else:
skipcount = skipcount + 1
print("該当ファイル数: {}".format(len(slices)))
print("スライス位置がないファイル: {}".format(skipcount))
# スライスの順番をそろえる
slices = sorted(slices, key=lambda s: s.SliceLocation) # .SliceLocationで場所情報を取得
# len(slices) = 270
# アスペクト比を計算する
ps = slices[0].PixelSpacing # ps = [0.571, 0.571] 1ピクセルの [y, x] 長さ
ss = slices[0].SliceThickness # ss = "3.0" スライスの厚み
ax_aspect = ps[0]/ps[1] # yの長さ/xの長さ = 1
sag_aspect = ss/ps[0] # スライスの厚み/y軸方向への1ピクセルの長さ = 3/0.571 = 5.25
cor_aspect = ss/ps[1] # スライスの厚み/x軸方向への1ピクセルの長さ = 3/0.571 = 5.25
# 空の3Dのnumpy arrayを作成する
img_shape = list(slices[0].pixel_array.shape) # img_shape = [512, 512]
img_shape.insert(0,len(slices)) # img_shape = [270, 512, 512]
img3d = np.zeros(img_shape) # 空のarrayを作る np.zeros([270, 512, 512])
# 3Dのnumpy arrayを作る
for i, s in enumerate(slices):
img2d = s.pixel_array # img2d.shape = (512, 512)
img3d[i,:, :] = img2d # img3d.shape = (270, 512, 512)
# プロットする
middle0 = img_shape[0]//2 #3d array の(z軸)頭尾軸中央 270/2 = 135
middle1 = img_shape[1]//2 #3d array の(y軸)上下軸中央 512/2 = 256
middle2 = img_shape[2]//2 #3d array の(x軸)左右軸中央 512/2 = 256
a1 = plt.subplot(1, 3, 1)
plt.imshow(img3d[middle0,:, :])
a1.set_aspect(ax_aspect) # ax_aspect = 1
a2 = plt.subplot(1, 3, 2)
plt.imshow(img3d[:, middle1, :])
a2.set_aspect(cor_aspect) # cor_aspect = 5.25
a3 = plt.subplot(1, 3, 3)
plt.imshow(img3d[:, :, middle2]) #
a3.set_aspect(sag_aspect) # sag_aspect = 5.25
plt.show()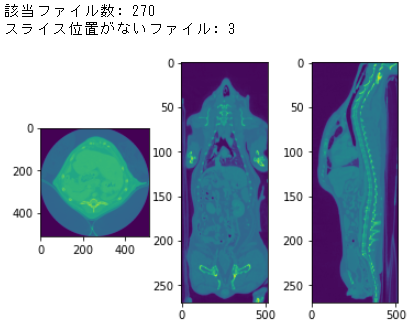
色が変だけど、とりあえず描写できました。
Numpyのarrayが理解できないので実験
numpy.ndarrayは以下のような構造をしています。
(x)→(y,x)→(z, y, x)
np.zeros([2,3,4])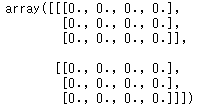
np.zeros([3,4,5])
array3d = np.array([[[1, 2, 3],
[4, 5, 6]],
[[7, 8, 9],
[10, 11, 12]],
[[13, 14, 15],
[16, 17, 18]]])
array3d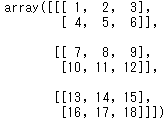
numpy.ndarrayは、図にしてみると以下のように理解することができます。
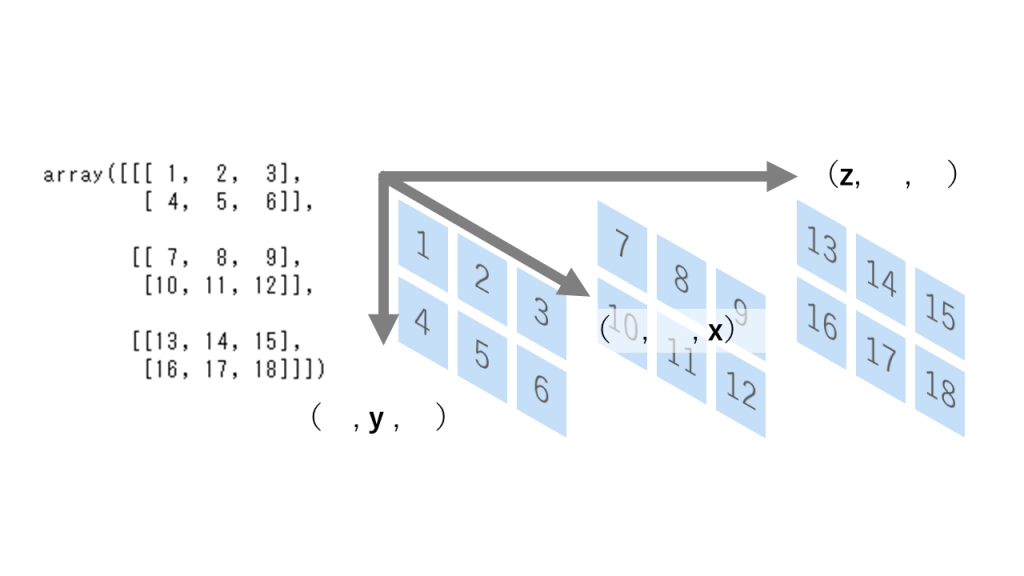
array3d[:,:,1]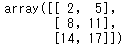
array3d[:,1,:]
array3d[1,:,:]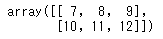
numpy.ndarrayの構造が分かったところで、実際の3Darrayを見てみることにします。
頭尾方向の中央のスライスを見てみます。
img3d[135, :, :]
matplotlibで表示してみたいと思います。
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[135, :, :])
a1.set_aspect(ax_aspect)
plt.show()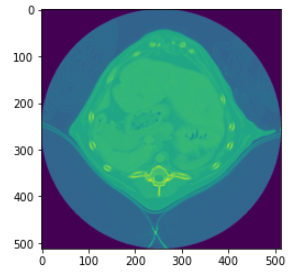
色が変なので、cmap = plt.cm.grayを追加します。
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[135, :, :], cmap = plt.cm.gray)
a1.set_aspect(ax_aspect)
plt.show()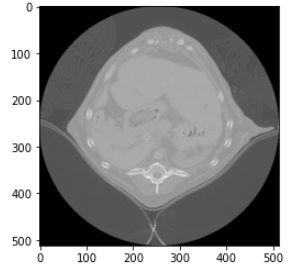
見慣れた色になってきました。コントラストを調整したいと思います。
vmin vmaxを追加してみます。
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[135, :, :], cmap=plt.cm.gray, vmin = 100, vmax = 300)
a1.set_aspect(ax_aspect)
plt.show()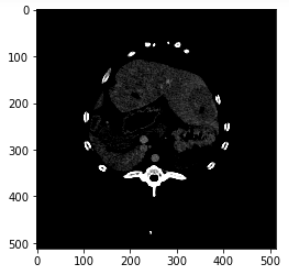
骨だけ抽出できました。vmin vmaxを変えてみます。
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[135, :, :], cmap=plt.cm.gray, vmin = 0, vmax =75)
a1.set_aspect(ax_aspect)
plt.show()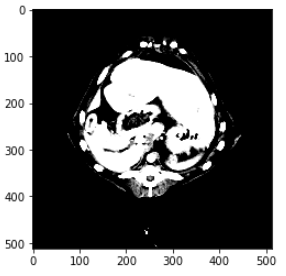
CT値は色々な臓器によって異なるので、目的の臓器のCT値に合わせてvminとvmaxを変化させるといいと思います。CTを撮影するマシンによって変わると思いますが、調べてみると以下がCT値の例となっていました。
| 骨 | 300 ~ 1000 |
| 軟部 | 30 ~ 50 |
| 脂肪 | -50 ~ -100 |
| 空気 | -1000 |
データのCT値について調べてみます。
CT値の最低は-2048、最高は1233ということがわかりました。
np.amin(img3d[135,:,:])-2048.0
np.amax(img3d[135,:,:])1233.0
どこの場所か確認してみます。
np.argmax(img3d[239,:,:])187148
これだとどこの場所だかわからないので、unravel_indexをします。
from numpy import unravel_index
unravel_index(np.argmax(img3d[135,:,:]), img3d[135,:,:].shape)(365, 268)
この付近を見てみたいと思います。
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[135,325:405,228:308], cmap=plt.cm.gray)
a1.set_aspect(ax_aspect)
plt.show()
このスライスでCT値が一番高いところは、脊椎ですね。
スライダーを使ってスライスの位置を移動させる
スライダーを使うことで、前後のスライスを次々とみることができます。
%matplotlib inline
import matplotlib.pyplot as plt
from ipywidgets import interact
import numpy as np
param = len(slices)
def f(k):
a1 = plt.subplot(1, 1, 1)
plt.imshow(img3d[k, :, :], cmap=plt.cm.gray, vmin = -500, vmax = 500)
a1.set_aspect(ax_aspect)
plt.show()
interact(f, k=(0,param-1) )次は、MPRのcoronal planeでスライダーを使ってみたいと思います。HPの都合上、静止画となっていますが、実際はスライダーを動かすことができます。
%matplotlib inline
import matplotlib.pyplot as plt
from ipywidgets import interact
import numpy as np
param = len(slices)
def f(k):
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(111)
plt.imshow(img3d[:, k, :], cmap=plt.cm.gray, vmin = -500, vmax = 500)
ax.set_aspect(sag_aspect)
plt.show()
interact(f, k=(0,param-1) )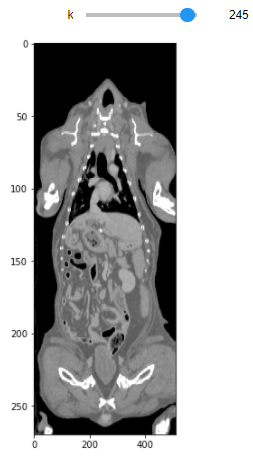
次は、MPRのsagital planeでスライダーを使ってみたいと思います。縦だとわかりにくいので、横に倒します。img3d[:, :, k].Tを使ってarrayを反転させます。それに伴い、アスペクト比も反転させます。
%matplotlib inline
import matplotlib.pyplot as plt
from ipywidgets import interact
import numpy as np
param = len(slices)
def f(k):
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(111)
plt.imshow(img3d[:, :, k].T, cmap=plt.cm.gray, vmin = -500, vmax = 500) # .T はtransposeで形を反転
ax.set_aspect(1/sag_aspect) # aspectを反転させます。
plt.show()
interact(f, k=(0,param-1) )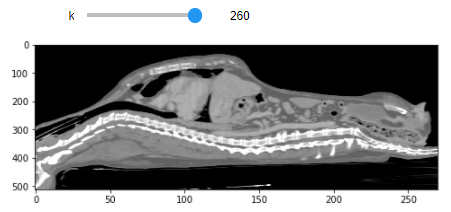
ところで、空気が一番CT値が低く-1000になるはず(勘違いポイント)なのに、このデータでは最低の-2048なのはなぜなのでしょうか。
CT値は英語でHounsfield Unitsです。
DICOMから直接取り出せるのは、pixelのデータであってCT値ではないようです。
CT値を得るには、以下の式で変換する必要があります。
Hounsfield Units = (pixel value) × (rescale slope) + rescale intercept
つまり今まで使用していたのpixel_arrayの数値は、CT値ではないということがわかりました。CT値を得るには、変換が必要です。
rescale slope と、rescale interceptの情報もDICOMファイルに含まれているので取得して、計算しなおします。
slices[0].RescaleSlope“1.0”
slices[0].RescaleIntercept“0.0”
Hounsfield Units = (pixel value) × 1 + 0
結局、傾きが1、切片が0なので、私の犬のCTデータは、pixel value は CT値と同じだということがわかりました。計算しなおさないで済みました。
では、どうして最低値が-2048なのでしょうか。
CTはキャリブレーションといって、基準となる水と空気をスキャンしたデータを使ってCT値の位置合わせを行っています。このCTは、キャリブレーション実施してあるはずなので空気は-1000あたりになるはずです。
また、2048は2の11乗です。CT値を-2000以下まで計測したいので、bitのキリがいいところ、-2048だと思います。1000以下まで計測したいなら、2の10乗で1024になったはずです。2048に特に意味はなく最低値を表示したというところでしょう。
ひょっとして、空気が最低値じゃないかもしれません。空気の数値を確認してみます。
plotlyを使ってインタラクティブに操作
plotlyを使うことでマウスホバーしたところのCT値が表示されるようになります。これもHPの都合上、静止画を貼っていますが、実際はインタラクティブです。空気のCT値はいくつか確認したいです。
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :])
fig.show()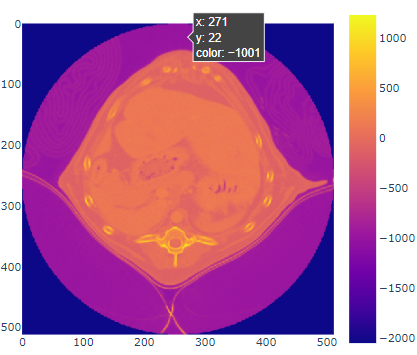
カーソルをプロット上に持っていくとCT値(color)を教えてくれるようになりました。空気はやはり-1000付近!-2028はどこでしょうか。
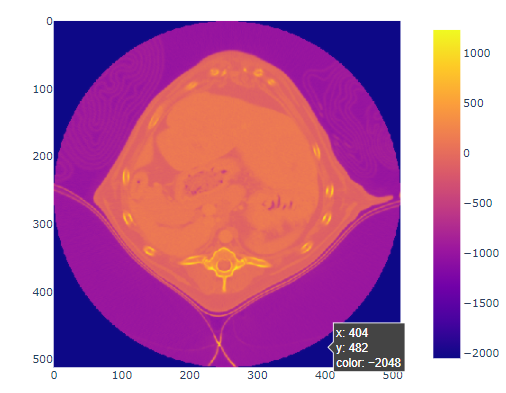
撮影円の外側が-2048。つまり、CTの撮影円の範囲外が-2048になっているだけでした。意外な盲点!!
CT値の最低値は、空気じゃなくて撮影範囲外です。
| 骨 | 300 ~ 1000 |
| 軟部 | 30 ~ 50 |
| 脂肪 | -50 ~ -100 |
| 空気 | -1000 |
| 撮影範囲外 | -2048 |
今度は、ちょうどよい色を探求したいと思います。
選択できる色(https://plotly.com/python/builtin-colorscales/)
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :], binary_string=True)
fig.show()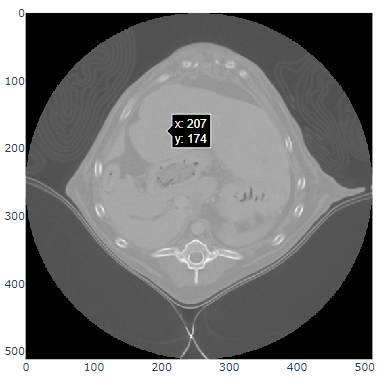
binary_string=Trueにすると、マウスホバーしてもピクセル値を教えてくれなくなります。
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :],color_continuous_scale='RdBu_r')
#fig = px.imshow(img3d[middle0,:, :],color_continuous_scale='RdBu') # _r reverse
fig.show()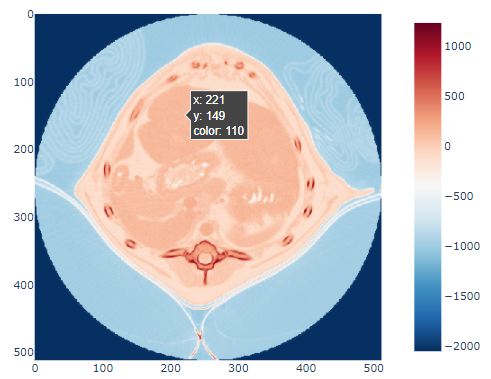
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :],color_continuous_scale='jet')
fig.show()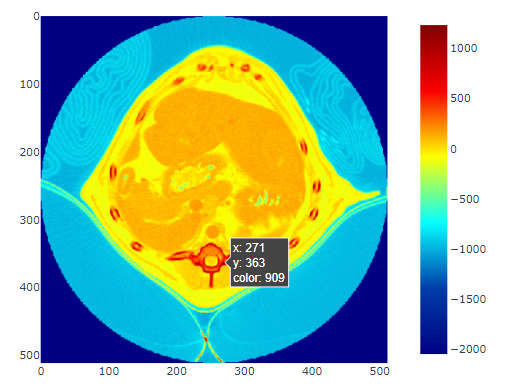
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :],color_continuous_scale='turbo')
fig.show()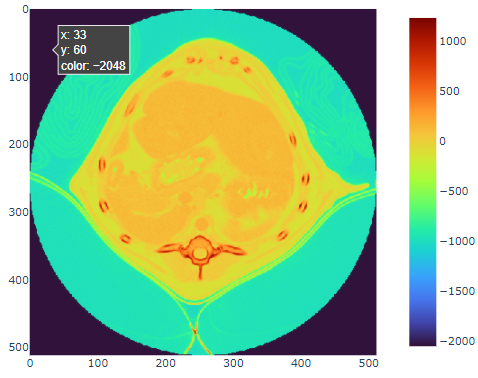
import plotly.express as px
fig = px.imshow(img3d[middle0,:, :],color_continuous_scale='thermal')
fig.show()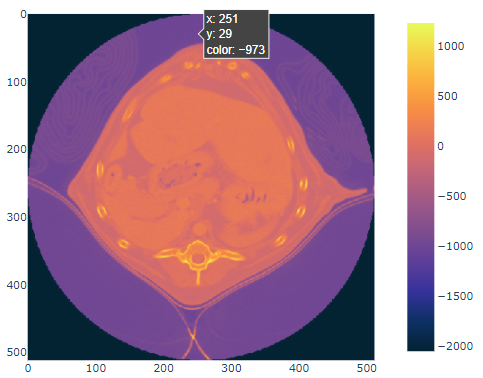
plotlyを使ってスライダー付きのインタラクティブなプロットを作成
スライスが多いと時間がかかるため、スライスを10枚に限定しています。
import plotly.express as px
fig = px.imshow(img3d[220:230,:,],
animation_frame=0,
labels=dict(animation_frame="slice"),
color_continuous_scale='jet')
fig.show()次回は、サーフェスレンダリングで骨をobjファイルとして保存してみます。
