dummyとonehotの違い
dummy encodingは、1列にデータにカテゴリが2つ(A、B)あると、AとBの列を作り、0か1を入れていき、どちらかの列を削除する。1列に3カテゴリであれば、3列(A、B、C)作り、1行削除し2列を作る。カテゴリn個ある場合は、n-1列で表現。
one hot encodingは、1列にデータにカテゴリが2つ(A、B)あると、AとBの列を作り、0か1を入れていく。1列に3カテゴリであれば、3列(A、B、C)作る。カテゴリn個ある場合は、n列で表現。
どちらを使うべきか
regressionから勉強するとdummyを使うべきと学びます。onehotはdummy trapにかかり、(他の列から1列を完璧に推測できてしまい)multicolinaryを起こしてしまう。逆に、neural network系から勉強するとdummyって何?って感じだと思います。
この本によると、linear regression models, logistic regression models, discriminant analysis, and Neural networks that don’t employ weight decayについては、ダミー変数化すべき。そのほかは、onehotで問題ない。ということらしい。
ただネットを調べてみるとneural networkについては両者でやってもあまり差がないので、単純なonehotの方が好まれるようです。weight decayを考慮したモデルはあまり見かけないと思います。
どちらかはっきりさせるために実験します。
① カテゴリをそのまま数字として扱ったケース
② dummy variable化して扱う
③ onehot encoding化して扱う
Autokeras(Neural Network)、Adaboost、Gradient Boost Machine、Random forest、Logistic regression、LDA、lightGBM(チューニング方法3通り)を使って、ACCURACYがどうなるかを見てみます。
データ
心臓病のデータセット。心臓病かどうかを各種データから予測する。
https://www.kaggle.com/cherngs/heart-disease-cleveland-uci
参考:https://archive.ics.uci.edu/ml/datasets/Heart+Disease
Authors:
1. Hungarian Institute of Cardiology. Budapest: Andras Janosi, M.D.
2. University Hospital, Zurich, Switzerland: William Steinbrunn, M.D.
3. University Hospital, Basel, Switzerland: Matthias Pfisterer, M.D.
4. V.A. Medical Center, Long Beach and Cleveland Clinic Foundation:Robert Detrano, M.D., Ph.D.
Outcome:
14列.condition: 0 = 心臓病なし, 1 = 心臓病あり category(nominal)
Predictors:
1列.age:年齢 numeric
2列.sex:性別 (1 = male; 0 = female) category(nominal)
3列.cp: 胸痛のタイプ (1: 典型的狭心症 2: 非典型的狭心症 3: 非狭心症痛 4: 無症状) category(nominal)
4列.trestbps:安静時血圧(入院時のmmHg単位)numeric
5列.chol: 血中コレストロール(mg/dl) numeric
6列.fbs:空腹時血糖値>120mg/dl(1=真、0=偽)category(nominal)
7列.restecg:安静時心電図結果 (0:正常 1:ST-T波異常(T波反転および/または0.05mV以上のST上昇もしくはST下降)を有する。 2:Estesの基準による左心室肥大の可能性が高いまたは確定しているもの)category(nominal)
8列.thalach:最大心拍数 numeric
9列.exang:運動誘発性狭心症(1=有、0=無)category(nominal)
10列.oldpeak:安静時に比べて運動により誘発されるSTの低下 numeric
11列.slope:運動負荷時のSTセグメントの傾き(1: upsloping 2: flat 3: downsloping)category(nominal)
12列.ca: X線透視検査で着色された主要血管の数(0-3)numeric
13列.thal:タリウム負荷試験の結果 0=正常、1=固定欠陥、2=可逆性欠陥 category(nominal)
コード
dummy encoding
pandasのget_dummiesを使う。drop_firstをTrueにする。
df = pd.read_csv("heart_cleveland_upload.csv")
dummys = pd.get_dummies(df,
drop_first = True,
columns=["sex","cp","fbs","restecg","exang","slope","thal","condition"])
onehot encoding
pandasのget_dummiesを使う。drop_firstをFalseにする。
df = pd.read_csv("heart_cleveland_upload.csv")
dummys = pd.get_dummies(df,
drop_first = False,
columns=["sex","cp","fbs","restecg","exang","slope","thal","condition"])
以下が10回、これらの条件を変化させてそれぞれのモデルでACCを計測した結果である。
① カテゴリをそのまま数字として扱ったケース(Unprocessed)
② dummy variable化して扱う (Dummy)
③ onehot encoding化して扱う (Onehot)
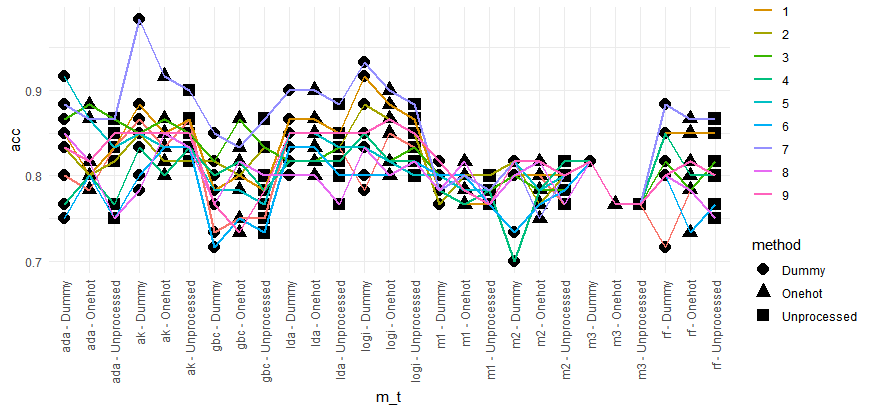
AutoKeras、logisticに関してはdummy encodingの方がよいスコアを出せそうです。(0.05で有意差はでませんでした)
逆に、Gradient Boost MachineのときはOnehotの方がよさそうです。
| 略 | 正式名 | Dummy mean ACC | Onehot mean ACC | Unprocessed mean ACC |
| ada | Adaboost | 0.835 | 0.823 | 0.816 |
| ak | Autokeras – Nerural Network | 0.855 | 0.845 | 0.848 |
| gbc | Gradient Boost Machine | 0.783 | 0.795 | 0.793 |
| lda | LDA | 0.84 | 0.84 | 0.83 |
| logi | Logistic regression | 0.851 | 0.841 | 0.836 |
| m1 | LightGBM optuna tuning method1 | 0.791 | 0.79 | 0.778 |
| m2 | LightGBM optuna tuning method2 | 0.79 | 0.786 | 0.791 |
| m3 | LightGBM without tuning | 0.816 | 0.766 | 0.766 |
| rf | Random Forest | 0.821 | 0.806 | 0.808 |
encodingの種類別のfor loop毎のACCのランク(ACCが高いほうがランク1となる)
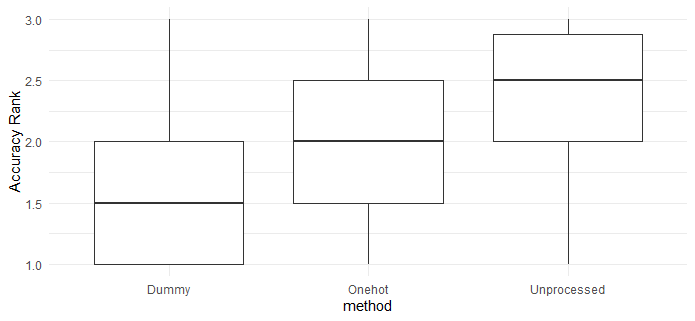
3群の平均ランキングの差 one-way ANOVA
9.54e-08 有意差あり
3群の平均ランキングの差 pairwise-t-test
| Dummy | Onehot | |
| Onehot | 0.00017 | |
| Unprocessed | 8.8e-08 | 0.08564 |
参考:コード
Onehotの時のコード
import seaborn as sns
import pandas as pd
acc_autokeras = []
acc_ada = []
acc_gbc = []
acc_rf = []
acc_logi = []
acc_lda = []
acc_lightgbm_m1 = []
acc_lightgbm_m2 = []
acc_lightgbm_m3 = []
for i in range(10):
import pandas as pd
from sklearn.model_selection import train_test_split as tts
df = pd.read_csv("heart_cleveland_upload.csv")
dummys = pd.get_dummies(df,
drop_first = False,
columns=["sex","cp","fbs","restecg","exang","slope","thal"])
train,test = tts(dummys,test_size=0.2,random_state=i)
x_train = train.drop("condition", axis=1).to_numpy()
y_train = train.loc[:,["condition"]].to_numpy().ravel()
x_test = test.drop("condition", axis=1).to_numpy()
y_test = test.loc[:,["condition"]].to_numpy().ravel()
import autokeras as ak
maxacc = 0
for i in range(5):
clf = ak.StructuredDataClassifier(overwrite=False, max_trials=30)
history = clf.fit(x_train, y_train, epochs=30,validation_split=0.2)
result = clf.evaluate(x_test, y_test)
print(result)
if result[1] > maxacc:
model_autokeras = clf.export_model()
model_autokeras.save("autokeras",save_format="tf")
maxacc = result[1]
print("saved",result)
else:
continue
from tensorflow.keras.models import load_model
from sklearn.metrics import accuracy_score
import numpy as np
# 重みの復元
manual = load_model("autokeras")
print("manual",manual.evaluate(x_test, y_test))
t = (manual.predict(x_test)>0.5)*1
flat_list = []
for sublist in t:
for item in sublist:
flat_list.append(item)
print(accuracy_score(np.array(flat_list),y_test))
acc_autokeras.append(accuracy_score(np.array(flat_list),y_test))
from sklearn.ensemble import RandomForestClassifier
clf_rf = RandomForestClassifier()
clf_rf.fit(x_train, y_train)
acc_rf.append(accuracy_score(clf_rf.predict(x_test),y_test))
from sklearn.ensemble import GradientBoostingClassifier
clf_gbc = GradientBoostingClassifier()
clf_gbc.fit(x_train, y_train)
acc_gbc.append(accuracy_score(clf_gbc.predict(x_test),y_test))
from sklearn.ensemble import AdaBoostClassifier
clf_ada = AdaBoostClassifier()
clf_ada.fit(x_train, y_train)
acc_ada.append(accuracy_score(clf_ada.predict(x_test),y_test))
from sklearn.linear_model import LogisticRegression
clf_logi = LogisticRegression(random_state=0,solver='liblinear')
clf_logi.fit(x_train, y_train)
acc_logi.append(accuracy_score(clf_logi.predict(x_test),y_test))
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
clf_lda = LinearDiscriminantAnalysis()
clf_lda.fit(x_train, y_train)
acc_lda.append(accuracy_score(clf_lda.predict(x_test),y_test))
############## creation of data for lightgbm ##################
from sklearn.model_selection import train_test_split as tts
import lightgbm as lgb
import pandas as pd
train_validation,test = tts(dummys,test_size=0.2,random_state=i)
train,validation = tts(train_validation,test_size=0.2,random_state=i)
x_train_validation = train_validation.drop("condition",axis=1).to_numpy()
y_train_validation = train_validation.loc[:,["condition"]].to_numpy().ravel()
x_train = train.drop("condition",axis=1).to_numpy()
y_train = train.loc[:,["condition"]].to_numpy().ravel()
x_val = validation.drop("condition",axis=1).to_numpy()
y_val = validation.loc[:,["condition"]].to_numpy().ravel()
x_test = test.drop("condition",axis=1).to_numpy()
y_test = test.loc[:,["condition"]].to_numpy().ravel()
lgb_train_validation = lgb.Dataset(x_train_validation,y_train_validation,free_raw_data=False)
lgb_train = lgb.Dataset(x_train, y_train,free_raw_data=False)
lgb_eval = lgb.Dataset(x_val, y_val,free_raw_data=False)
############ method 1 ##############
import optuna
import numpy as np
import sklearn
def objective(trial):
param = {
'objective': 'binary',
'metric': trial.suggest_categorical('metric', ['binary_error',"binary_logloss"]),
'lambda_l1': trial.suggest_loguniform('lambda_l1', 1e-8, 10.0),
'lambda_l2': trial.suggest_loguniform('lambda_l2', 1e-8, 10.0),
'num_leaves': trial.suggest_int('num_leaves', 2, 256),
'feature_fraction': trial.suggest_uniform('feature_fraction', 0.4, 1.0),
'bagging_fraction': trial.suggest_uniform('bagging_fraction', 0.4, 1.0),
'bagging_freq': trial.suggest_int('bagging_freq', 1, 7),
'min_child_samples': trial.suggest_int('min_child_samples', 5, 100),
}
gbm = lgb.train(param, lgb_train,valid_sets=lgb_eval)
preds = gbm.predict(x_val)
pred_labels = np.rint(preds)
accuracy = sklearn.metrics.accuracy_score(y_val, pred_labels)
return accuracy
study = optuna.create_study(direction='maximize')
study.optimize(objective, n_trials=1000)
bestparams = study.best_trial.params
bestparams["objective"]="binary"
model = lgb.train(bestparams, lgb_train_validation)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method1",bestparams)
print(sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m1.append(sklearn.metrics.accuracy_score(pred,y_test))
############ method 1 ##############
############ method 2 ##############
import optuna.integration.lightgbm as LGB_optuna
param = {
'objective': 'binary',
'metric': 'binary_error',
}
lgb_train = lgb.Dataset(x_train, y_train,free_raw_data=False)
lgb_eval = lgb.Dataset(x_val, y_val,free_raw_data=False)
best = LGB_optuna.train(param, lgb_train,valid_sets=lgb_eval)
model = lgb.train(best.params, lgb_train_validation)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method2",best.params )
print( sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m2.append(sklearn.metrics.accuracy_score(pred,y_test))
################ method 2 ######################
################ method 3 ######################
param = {
'objective': 'binary',
'metric': 'binary_error',
}
model = lgb.train(param, lgb_train,valid_sets=lgb_eval)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method3",model.params)
print(sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m3.append(sklearn.metrics.accuracy_score(pred,y_test))
pd.DataFrame({"ak":acc_autokeras,
"ada":acc_ada,
"gbc":acc_gbc,
"rf":acc_rf,
"logi":acc_logi,
"lda":acc_lda,
"m1":acc_lightgbm_m1,
"m2":acc_lightgbm_m2,
"m3":acc_lightgbm_m3,
}).to_csv("data_with_onehot.csv")