動機
病気の自動診断装置的なものを、機械学習(AI)を使って作りたいと思います。データさえあれば色々できると思いますが、公開されているデータはあまりないように思います。そういった中でも心臓病のデータがあったので、自動診断の可能性について研究したいと思います。pythonとRを両方使っています。
データ
心臓病のデータセット。心臓病かどうかを各種データから予測する。
https://www.kaggle.com/cherngs/heart-disease-cleveland-uci
参考:https://archive.ics.uci.edu/ml/datasets/Heart+Disease
Authors:
1. Hungarian Institute of Cardiology. Budapest: Andras Janosi, M.D.
2. University Hospital, Zurich, Switzerland: William Steinbrunn, M.D.
3. University Hospital, Basel, Switzerland: Matthias Pfisterer, M.D.
4. V.A. Medical Center, Long Beach and Cleveland Clinic Foundation:Robert Detrano, M.D., Ph.D.
Outcome:
14列.condition: 0 = no disease, 1 = disease category(nominal)
Predictors:
1列.age:年齢 numeric
2列.sex:性別 (1 = male; 0 = female) category(nominal)
3列.cp: 胸痛のタイプ (1: 典型的狭心症 2: 非典型的狭心症 3: 非狭心症痛 4: 無症状) category(nominal)
4列.trestbps:安静時血圧(入院時のmmHg単位)numeric
5列.chol: 血中コレストロール(mg/dl) numeric
6列.fbs:空腹時血糖値>120mg/dl(1=真、0=偽)category(nominal)
7列.restecg:安静時心電図結果 (0:正常 1:ST-T波異常(T波反転および/または0.05mV以上のST上昇もしくはST下降)を有する。 2:Estesの基準による左心室肥大の可能性が高いまたは確定しているもの)category(nominal)
8列.thalach:最大心拍数 numeric
9列.exang:運動誘発性狭心症(1=有、0=無)category(nominal)
10列.oldpeak:安静時に比べて運動により誘発されるSTの低下 numeric
11列.slope:運動負荷時のSTセグメントの傾き(1: upsloping 2: flat 3: downsloping)category(nominal)
12列.ca: X線透視検査で着色された主要血管の数(0-3)numeric
13列.thal:タリウム負荷試験の結果 0=正常、1=固定欠陥、2=可逆性欠陥 category(nominal)
カテゴリ間にunbalanceがあるか確認します。グラフ化します。
import seaborn as sns
import pandas as pd
df = pd.read_csv("heart_cleveland_upload.csv")
described = df.describe()
edadf = df.loc[:,["sex","cp","fbs","restecg","exang","slope","thal","condition"]]
edadf = edadf.melt()
sns.set_palette("Set1")
sns.violinplot(x='variable', y='value', data=edadf)
sns.set_palette("Set2")
sns.swarmplot(x='variable', y='value', data=edadf,dodge=True)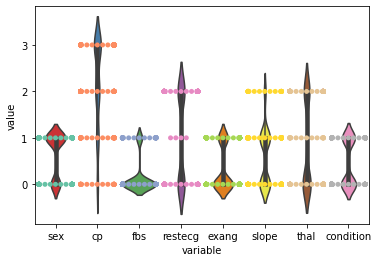
グラフ化するとfbsがあきらかに0に多い傾向があることがわかります。
targetのconditionが均等っぽいのでよしとします。
dummy encoding?onehot encoding?
変数の中には、カテゴリ変数があります。これらの数字の順序に意味はないので、変数の取り扱いに工夫しなければなりません。カテゴリ変数の取り扱いは、dummy encodingか、onehot encodingの2つが有名です。どちらを使った方がいいか、こちらの記事で検討しました。今回は、dummyを選択します。
今回の実験では以下が目的
Autokeras、adaboost、gradient boost machine、random forest、logistic regression、LDA、lightGBMを使って心臓病予測モデルを作成。
import seaborn as sns
import pandas as pd
acc_autokeras = []
acc_ada = []
acc_gbc = []
acc_rf = []
acc_logi = []
acc_lda = []
acc_lightgbm_m1 = []
acc_lightgbm_m2 = []
acc_lightgbm_m3 = []
for i in range(10):
import pandas as pd
from sklearn.model_selection import train_test_split as tts
df = pd.read_csv("heart_cleveland_upload.csv")
dummys = pd.get_dummies(df,
drop_first = True,
columns=["sex","cp","fbs","restecg","exang","slope","thal"])
train,test = tts(dummys,test_size=0.2,random_state=1)
x_train = train.drop("condition", axis=1).to_numpy()
y_train = train.loc[:,["condition"]].to_numpy().ravel()
x_test = test.drop("condition", axis=1).to_numpy()
y_test = test.loc[:,["condition"]].to_numpy().ravel()
import autokeras as ak
maxacc = 0
for i in range(5):
clf = ak.StructuredDataClassifier(overwrite=False, max_trials=30)
history = clf.fit(x_train, y_train, epochs=30,validation_split=0.2)
result = clf.evaluate(x_test, y_test)
print(result)
if result[1] > maxacc:
model_autokeras = clf.export_model()
model_autokeras.save("autokeras",save_format="tf")
maxacc = result[1]
print("saved",result)
else:
continue
from tensorflow.keras.models import load_model
from sklearn.metrics import accuracy_score
import numpy as np
# 重みの復元
manual = load_model("autokeras")
print("manual",manual.evaluate(x_test, y_test))
t = (manual.predict(x_test)>0.5)*1
flat_list = []
for sublist in t:
for item in sublist:
flat_list.append(item)
print(accuracy_score(np.array(flat_list),y_test))
acc_autokeras.append(accuracy_score(np.array(flat_list),y_test))
from sklearn.ensemble import RandomForestClassifier
clf_rf = RandomForestClassifier()
clf_rf.fit(x_train, y_train)
acc_rf.append(accuracy_score(clf_rf.predict(x_test),y_test))
from sklearn.ensemble import GradientBoostingClassifier
clf_gbc = GradientBoostingClassifier()
clf_gbc.fit(x_train, y_train)
acc_gbc.append(accuracy_score(clf_gbc.predict(x_test),y_test))
from sklearn.ensemble import AdaBoostClassifier
clf_ada = AdaBoostClassifier()
clf_ada.fit(x_train, y_train)
acc_ada.append(accuracy_score(clf_ada.predict(x_test),y_test))
from sklearn.linear_model import LogisticRegression
clf_logi = LogisticRegression(random_state=0,solver='liblinear')
clf_logi.fit(x_train, y_train)
acc_logi.append(accuracy_score(clf_logi.predict(x_test),y_test))
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
clf_lda = LinearDiscriminantAnalysis()
clf_lda.fit(x_train, y_train)
acc_lda.append(accuracy_score(clf_lda.predict(x_test),y_test))
############## creation of data for lightgbm ##################
from sklearn.model_selection import train_test_split as tts
import lightgbm as lgb
import pandas as pd
train_validation,test = tts(dummys,test_size=0.2,random_state=i)
train,validation = tts(train_validation,test_size=0.2,random_state=i)
x_train_validation = train_validation.drop("condition",axis=1).to_numpy()
y_train_validation = train_validation.loc[:,["condition"]].to_numpy().ravel()
x_train = train.drop("condition",axis=1).to_numpy()
y_train = train.loc[:,["condition"]].to_numpy().ravel()
x_val = validation.drop("condition",axis=1).to_numpy()
y_val = validation.loc[:,["condition"]].to_numpy().ravel()
x_test = test.drop("condition",axis=1).to_numpy()
y_test = test.loc[:,["condition"]].to_numpy().ravel()
lgb_train_validation = lgb.Dataset(x_train_validation,y_train_validation,free_raw_data=False)
lgb_train = lgb.Dataset(x_train, y_train,free_raw_data=False)
lgb_eval = lgb.Dataset(x_val, y_val,free_raw_data=False)
############ method 1 ##############
import optuna
import numpy as np
import sklearn
def objective(trial):
param = {
'objective': 'binary',
'metric': trial.suggest_categorical('metric', ['binary_error',"binary_logloss"]),
'lambda_l1': trial.suggest_loguniform('lambda_l1', 1e-8, 10.0),
'lambda_l2': trial.suggest_loguniform('lambda_l2', 1e-8, 10.0),
'num_leaves': trial.suggest_int('num_leaves', 2, 256),
'feature_fraction': trial.suggest_uniform('feature_fraction', 0.4, 1.0),
'bagging_fraction': trial.suggest_uniform('bagging_fraction', 0.4, 1.0),
'bagging_freq': trial.suggest_int('bagging_freq', 1, 7),
'min_child_samples': trial.suggest_int('min_child_samples', 5, 100),
}
gbm = lgb.train(param, lgb_train,valid_sets=lgb_eval)
preds = gbm.predict(x_val)
pred_labels = np.rint(preds)
accuracy = sklearn.metrics.accuracy_score(y_val, pred_labels)
return accuracy
study = optuna.create_study(direction='maximize')
study.optimize(objective, n_trials=1000)
bestparams = study.best_trial.params
bestparams["objective"]="binary"
model = lgb.train(bestparams, lgb_train_validation)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method1",bestparams)
print(sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m1.append(sklearn.metrics.accuracy_score(pred,y_test))
############ method 1 ##############
############ method 2 ##############
import optuna.integration.lightgbm as LGB_optuna
param = {
'objective': 'binary',
'metric': 'binary_error',
}
lgb_train = lgb.Dataset(x_train, y_train,free_raw_data=False)
lgb_eval = lgb.Dataset(x_val, y_val,free_raw_data=False)
best = LGB_optuna.train(param, lgb_train,valid_sets=lgb_eval)
model = lgb.train(best.params, lgb_train_validation)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method2",best.params )
print( sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m2.append(sklearn.metrics.accuracy_score(pred,y_test))
################ method 2 ######################
################ method 3 ######################
param = {
'objective': 'binary',
'metric': 'binary_error',
}
model = lgb.train(param, lgb_train,valid_sets=lgb_eval)
pred = model.predict(x_test)
pred = (pred > 0.5) * 1
print("method3",model.params)
print(sklearn.metrics.accuracy_score(pred,y_test))
acc_lightgbm_m3.append(sklearn.metrics.accuracy_score(pred,y_test))
pd.DataFrame({"ak":acc_autokeras,
"ada":acc_ada,
"gbc":acc_gbc,
"rf":acc_rf,
"logi":acc_logi,
"lda":acc_lda,
"m1":acc_lightgbm_m1,
"m2":acc_lightgbm_m2,
"m3":acc_lightgbm_m3,
}).to_csv("data_with_dummy.csv")
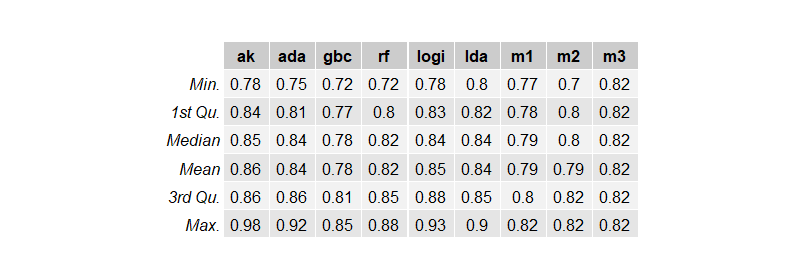
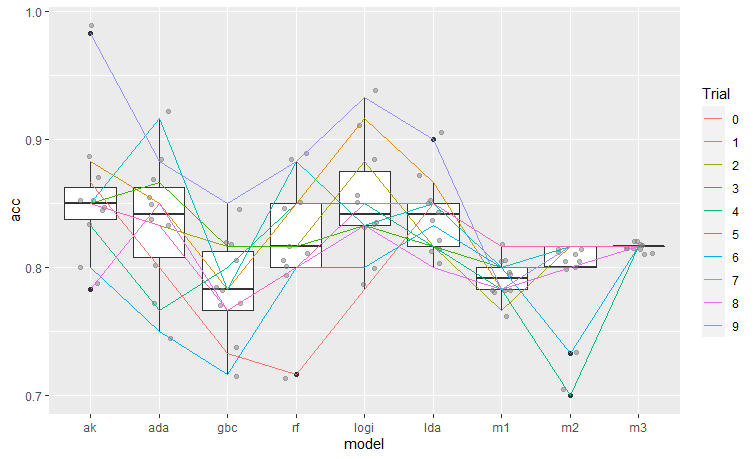
Autokerasが一番よさそうです。ACCが86%になりました。
でも、Neural NetworkだとFeature Importanceが見れないので研究のためにadaboost、logistic regressionを使ってみたいと思います。logistic regressionはfeature importanceを見るために(pythonだと)大変な思いをしますのでadaboostにしておきます。
まずはAdaboostでAccuracyとfeature importanceを見てみます。
import pandas as pd
from sklearn.metrics import accuracy_score
df = pd.read_csv("heart_cleveland_upload.csv")
dummys = pd.get_dummies(df,
drop_first = True,
columns=["sex","cp","fbs","restecg","exang","slope","thal"])
train,test = tts(dummys,test_size=0.2,random_state=15)
x_train = train.drop("condition", axis=1)
y_train = train.loc[:,["condition"]]
x_test = test.drop("condition", axis=1)
y_test = test.loc[:,["condition"]]
from sklearn.ensemble import AdaBoostClassifier
clf_ada = AdaBoostClassifier()
clf_ada.fit(x_train, y_train.to_numpy().ravel())
accuracy_score(clf_ada.predict(x_test),y_test)
importance = clf_ada.feature_importances_
resultdf = pd.DataFrame(columns = ["feature","importance"],
index= range(len(x_train.columns)))
for i,feat in enumerate(x_train.columns):
resultdf.iloc[i,0] = feat
resultdf.iloc[i,1] = importance[i]
import seaborn as sns
import matplotlib.pyplot as plt
sns.barplot(x=resultdf.iloc[:,0],y=resultdf.iloc[:,1])
plt.xticks(rotation = 90)accuracy_score(clf_ada.predict(x_test),y_test)
Out[145]: 0.783
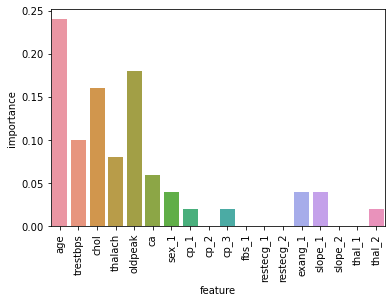
age、oldpeak、cholが大事なトップ3でした。この3つだけの情報しかとれない状況で、3変数のみで再学習・推測させた場合にはスコアはどうなるでしょうか。
x_train = train.drop("condition", axis=1)
x_train2 = x_train.loc[:,["age","oldpeak","chol"]]
y_train = train.loc[:,["condition"]]
x_test = test.drop("condition", axis=1)
x_test2 = x_test.loc[:,["age","oldpeak","chol"]]
y_test = test.loc[:,["condition"]]
clf_ada = AdaBoostClassifier()
clf_ada.fit(x_train2, y_train.to_numpy().ravel())
accuracy_score(clf_ada.predict(x_test2),y_test)accuracy_score(clf_ada.predict(x_test2),y_test)
Out[147]: 0.65
ACCは0.1ほど低下しました。例えば、この3つの質問だけしか聞けなかった場合はどうしたらよいでしょうか。かつ、学習モデルはサーバー上にあるために再学習することはできずに、他の変数についてもデータを与える必要がある場合にはどうしたらよいでしょうか。
その場合には他の質問は、推測してimputationするしかなさそうです。imputationの仕方は様々ありますが、平均値を入れるもしくは、マジョリティのクラスを入れる方法が一般的です。やってみたいと思います。(Neural Networkならmask layerを入れることで実現できそうです)
x_test = x_test.reset_index(drop=True)
x_test3 = pd.DataFrame(columns = x_test.columns,
index = range(len(x_test)))
x_test3.loc[:,["age","oldpeak","chol"]] = x_test.loc[:,["age","oldpeak","chol"]]
for i in range(len(x_test.columns)):
print(x_test.columns[i])
if x_test.columns[i] in ["age","oldpeak","chol"]:
continue
else:
if any(x_test.iloc[:,i] > 5):
print("mean", x_test.iloc[:,i].mean())
x_test3[x_test.columns[i]] = x_test.iloc[:,i].mean()
else:
print("count\n",x_test.iloc[:,i].value_counts())
temp = x_test.iloc[:,i].value_counts()
x_test3[x_test.columns[i]] = temp.index[np.argmax(temp)]
y_test3 = test.loc[:,["condition"]]
accuracy_score(clf_ada.predict(x_test3),y_test3)accuracy_score(clf_ada.predict(x_test3),y_test3)
Out[219]: 0.566
ACCが0.56まで下がってきてしまいました。やはり3変数だけにして再学習させて推測させたほうがいいスコアになります。
Logistic Regressionを研究。
Rでは、caretというパッケージでlogistic regressionにおいてもfeature importance (variable importance)を計算してくれます。その計算方法は、z値の絶対値のようです。z値 = coef / std err
pythonのsklearnのlogistic regressionではcoefはでますがz値は出ないようです。そのためstatsmodels.apiを使用する必要があります。まずは、sklearnとstatsmodelのlogistic regressionのcoefが同じことを確認します。
from sklearn.linear_model import LogisticRegression
clf_logi = LogisticRegression(solver='liblinear')
clf_logi.fit(x_train, y_train.to_numpy().ravel())
clf_logi.coef_
accuracy_score(clf_logi.predict(x_test),y_test)Out[231]:
array([[-0.04193568, 0.01651299, 0.00271643, -0.0259583 , 0.42127073,
1.0348819 , 0.80445348, 0.58375026, -0.31518106, 1.25910402,
-0.15095486, 0.12876544, 0.5238023 , 0.48165847, 0.86682758,
0.02635701, 0.15345965, 1.28749637]])
accuracy_score(clf_logi.predict(x_test),y_test)
Out[232]: 0.9
import statsmodels.api as sm
log_reg = sm.Logit(y_train, x_train).fit()
pred = log_reg.predict(x_train)
log_reg.summary()
accuracy_score((log_reg.predict(x_test)>0.5)*1,y_test)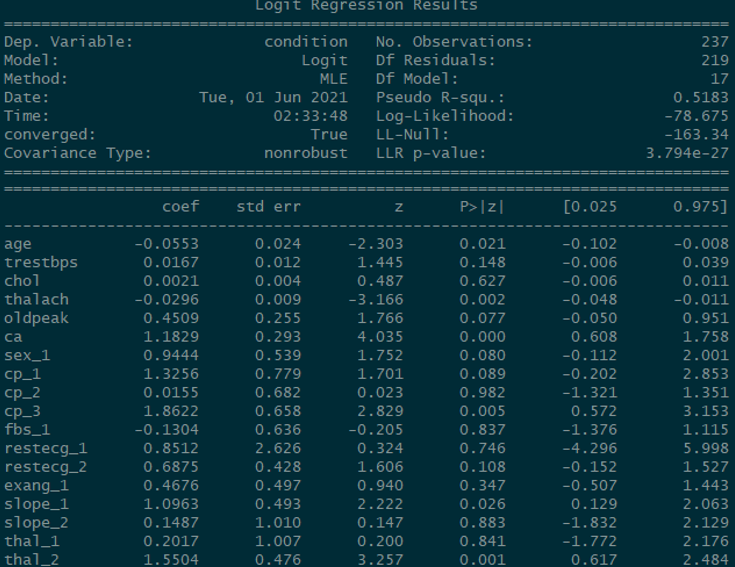
accuracy_score((log_reg.predict(x_test)>0.5)*1,y_test)
Out[236]: 0.883
ACCも違えば、coefも違う。。ACCが違うのは気持ち悪い。ひょっとしてROC
でカットオフ値を学習させたらSklearnのACCに近づくかも?
import statsmodels.api as sm
import numpy as np
from sklearn import metrics
log_reg = sm.Logit(y_train, x_train).fit()
pred = log_reg.predict(x_train)
fpr, tpr, thresholds = metrics.roc_curve(y_train,pred)
bestthresh = thresholds[np.argmax(tpr - fpr)]
predclass = (log_reg.predict(x_test)>bestthresh)*1
accuracy_score(predclass, y_test)Out[237]: 0.866
ACC下がってしまった。やる気がなくなってきました。Rでやってみます。
train.to_csv("train.csv")
test.to_csv("test.csv")train_df <- read.csv("train.csv")
test_df <- read.csv("test.csv")
logistic <- glm(condition~.,data=train_df,family=binomial)
pred <- predict(logistic,
newdata = test_df,
type = "response")
table( prediction = (pred > 0.5)*1,
true = test_df$condition)
(28+25)/(25+25+3+4)ACCは0.92 でした。
library(caret)
varImp.lg <- varImp(logistic)
ggplot(data= varImp.lg, aes(x=rownames(varImp.lg),y=Overall)) +
geom_bar(position="dodge",stat="identity",width = 0, color = "black") +
coord_flip() + geom_point(color='skyblue') + xlab(" Importance Score")+
ggtitle("Variable Importance") +
theme(plot.title = element_text(hjust = 0.5)) +
theme(panel.background = element_rect(fill = 'white', colour = 'black'))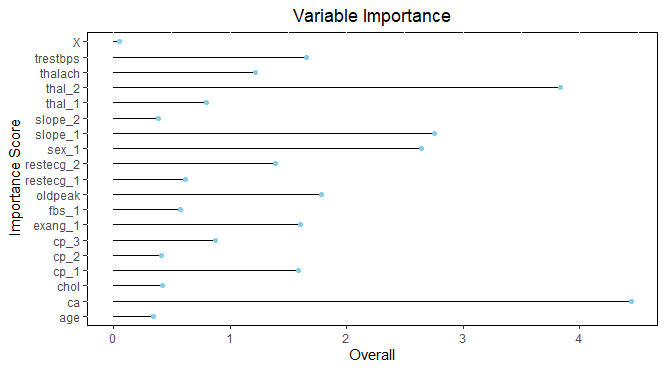
さきほどのADAboostとは全く異なる結果になりました。
データをtrain test splitする際のシードによって変数の重要度が毎回異なるので、複数回実施して、安定的に上位にくる変数が重要であると結論したほうがよさそうです。1回だけの試行で、変数の重要度を決めるのは危険です。
複数回実行して、変数の重要度を最終決定するのがベストですが、力尽きました。
こちらの記事で、変数の重要度を決める試行を行っています。
RでのLogistic regressionの記事はこちらです。
